2021.10.25
- プレスリリース
- 研究
DNAの1塩基の違いをハイスループットに判別できる新技術「PRIMA」を開発 −ゲノム編集個体を迅速・簡便・安価に選抜−
横浜市立大学木原生物学研究所 角井宏行 特任助教(研究当時、現 新潟大学 特任助教)、清水健太郎 客員教授(チューリッヒ大学教授兼任)、チューリッヒ大学 山﨑美紗子 博士の研究グループは、DNAの1塩基の違いを電気泳動法により効率的に判別できる新しい手法PRIMA(プリマ, Probe-induced Heteroduplex Mobility Assay)を開発しました。本手法は、5塩基欠失をもつ40塩基の一本鎖DNAをプローブ*1として用いることで、従来の電気泳動法では見分けることが難しかったDNAの1塩基というわずかな違いを迅速・簡便・安価に判別できます。1塩基挿入/欠失個体を効率的に判別できるだけでなく、塩基のタイプの違い(一塩基多型: SNP*2)を判別できる事例も示し、現在盛んに行われているゲノム編集*3研究やゲノムDNA研究、遺伝子多型解析をさらに加速させることができると期待されます。
本研究成果は、「Scientific Reports誌」に掲載されました。(日本時間2021年10月24日18時)また、本技術は横浜市立大学とチューリッヒ大学による国際特許を出願中です。
本研究成果は、「Scientific Reports誌」に掲載されました。(日本時間2021年10月24日18時)また、本技術は横浜市立大学とチューリッヒ大学による国際特許を出願中です。
研究成果のポイント
|
研究背景
近年のゲノム編集技術の発達により、さまざまな生物で標的DNA配列特異的に変異を導入することが容易になってきています。ゲノム編集によってDNA二本鎖切断が生じ、非相同末端結合によって修復されたDNA配列の多くは1塩基の挿入もしくは欠失の変異を有することがわかっています。この1塩基挿入/欠失変異はタンパク質中のアミノ酸の変異を引き起こすため、標的遺伝子の破壊を行うという点では望ましい変異のタイプであるものの、わずか“DNA 1塩基の差”という微細な違いを見分けるには、DNAシークエンスによる塩基配列の確認などが必要であるため、多くのサンプルの解析にはコストや時間が必要でした。
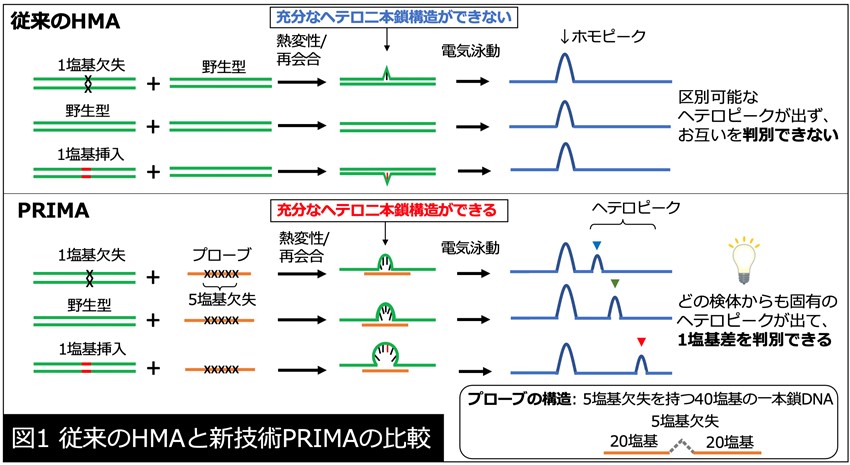
電気泳動法の一つであるHMA(Heteroduplex Mobility Assay, ヘテロ二本鎖移動度分析)は野生型と長さの異なる変異型のDNA断片を結合させたときに、ヘテロ二本鎖構造*4を取り、その移動度が遅くなることで変異型配列が存在することを判別する手法です。HMAはシークエンスと比較して非常に簡便で安価な手法ですが、DNAの1塩基の違いでは充分なヘテロ二本鎖構造ができないため、従来のHMAでは1塩基挿入/欠失配列を見分けることは困難でした(図1上)。
電気泳動法の一つであるHMA(Heteroduplex Mobility Assay, ヘテロ二本鎖移動度分析)は野生型と長さの異なる変異型のDNA断片を結合させたときに、ヘテロ二本鎖構造*4を取り、その移動度が遅くなることで変異型配列が存在することを判別する手法です。HMAはシークエンスと比較して非常に簡便で安価な手法ですが、DNAの1塩基の違いでは充分なヘテロ二本鎖構造ができないため、従来のHMAでは1塩基挿入/欠失配列を見分けることは困難でした(図1上)。
研究内容
私たちは野生型と1塩基だけの挿入/欠失のDNA断片の組み合わせではヘテロ二本鎖構造ができないことを確認した一方で、野生型と4〜6塩基欠失との組み合わせでは充分なヘテロ二本鎖構造ができることに注目しました。つまり、従来通り野生型と同じ配列をサンプルと反応させるのではなく、最初から5塩基を欠失した配列を“プローブ”としてサンプルと反応させることで、1塩基欠失配列とは4塩基差、野生型配列とは5塩基差、1塩基挿入配列とは6塩基差を作り出し、充分なヘテロ二本鎖構造を生じさせることができると考えました(図1下)。実際に5塩基欠失配列をプローブとして用いて野生型と1塩基挿入/欠失配列を解析したところ、それぞれのサンプルから固有のサイズをもつヘテロピークを検出でき、それぞれの配列を明確に判別できることがわかりました(図2)。この5塩基欠失プローブは40塩基の一本鎖DNAで機能し、数百円程度で簡単に入手することができます。これらの工夫を行うことでDNAの1塩基差を迅速・簡便・安価に判別できる手法として確立し、PRIMA(プリマ)と名付けました(図1下)。
私たちはこれまでに植物やバクテリア、ヒトの配列を標的にしてPRIMAを行い、1塩基挿入/欠失を見分けることができることを確認しており、PRIMAはシンプルなプロトコル(図3)でありつつも様々な生物種の配列に広く適用できるパワフルな手法であると考えています。さらに、PRIMAにより、DNAの長さは全く同じで塩基の種類(A/T/G/C)だけが異なる一塩基多型(SNP)についてもマイクロチップ電気泳動によって判別できる例を示し、SNPタイピングができる可能性を示しました(図4)。
私たちはこれまでに植物やバクテリア、ヒトの配列を標的にしてPRIMAを行い、1塩基挿入/欠失を見分けることができることを確認しており、PRIMAはシンプルなプロトコル(図3)でありつつも様々な生物種の配列に広く適用できるパワフルな手法であると考えています。さらに、PRIMAにより、DNAの長さは全く同じで塩基の種類(A/T/G/C)だけが異なる一塩基多型(SNP)についてもマイクロチップ電気泳動によって判別できる例を示し、SNPタイピングができる可能性を示しました(図4)。
今後の展開
現在ゲノム編集による研究は生物種を超えて発展し続けており、PRIMAのような様々な配列に適用可能で迅速・簡便・安価にDNAの1塩基差を見分ける手法は今後さらに必要とされると考えられます。また、同種の個体間でゲノムDNAを比較すると、無数の1塩基挿入/欠失配列や1塩基多型が存在し、中には病気や重要な形質に影響を与える多型も存在します。PRIMAはこれらの多型をハイスループットに検出できるため、ゲノムDNA研究や医療、育種などに広く適用できる手法として、今後さまざまな研究分野で用いられることが期待できます。
研究費
本研究は、科学技術振興機構(JST)CREST「環境変動に対する植物の頑健性の解明と応用に向けた基盤技術の創出」、文部科学省科研費 新学術領域研究「植物新種誕生の原理」、スイス科学財団、日本学術振興会科学研究費助成事業などの支援を受けて実施されました。
論文情報
タイトル: PRIMA: a rapid and cost-effective genotyping method to detect single-nucleotide differences using probe-induced heteroduplexes
著者: Hiroyuki Kakui, Misako Yamazaki, Kentaro K. Shimizu
掲載雑誌: Scientific Reports
DOI: https://doi.org/10.1038/s41598-021-99641-x
著者: Hiroyuki Kakui, Misako Yamazaki, Kentaro K. Shimizu
掲載雑誌: Scientific Reports
DOI: https://doi.org/10.1038/s41598-021-99641-x
特許情報
PROBE-INDUCED HETERODUPLEX MOBILITY ASSAY
国際特許出願番号: PCT/EP2020/072434
https://patentscope2.wipo.int/search/en/detail.jsf?docId=WO2021023896&tab=PCTBIBLIO
国際特許出願番号: PCT/EP2020/072434
https://patentscope2.wipo.int/search/en/detail.jsf?docId=WO2021023896&tab=PCTBIBLIO
用語説明
*1 プローブ:
対象、標的を探るための道具。タンパク質を検出するための抗体やGFP等がこれにあたる。
*2 一塩基多型(Single Nucleotide Polymorphism, SNP):
同じ種の集団の中に存在するゲノム配列内の違いの中で、一塩基の違いをこう呼ぶ。読み方はスニップ。
*3 ゲノム編集技術:
人工のDNA切断システムを利用して、標的遺伝子のDNA配列を高い精度で編集・改変する技術。
*4 ヘテロ二本鎖構造:
部分的に異なるDNA配列のセンス鎖とアンチセンス鎖が結合した時にできるDNAの”ひずみ”。このひずみに充分な違いがあると電気泳動での移動度が遅くなるため、同一配列の二本鎖DNA(ホモ二本鎖)と区別することができる。
対象、標的を探るための道具。タンパク質を検出するための抗体やGFP等がこれにあたる。
*2 一塩基多型(Single Nucleotide Polymorphism, SNP):
同じ種の集団の中に存在するゲノム配列内の違いの中で、一塩基の違いをこう呼ぶ。読み方はスニップ。
*3 ゲノム編集技術:
人工のDNA切断システムを利用して、標的遺伝子のDNA配列を高い精度で編集・改変する技術。
*4 ヘテロ二本鎖構造:
部分的に異なるDNA配列のセンス鎖とアンチセンス鎖が結合した時にできるDNAの”ひずみ”。このひずみに充分な違いがあると電気泳動での移動度が遅くなるため、同一配列の二本鎖DNA(ホモ二本鎖)と区別することができる。
参考図
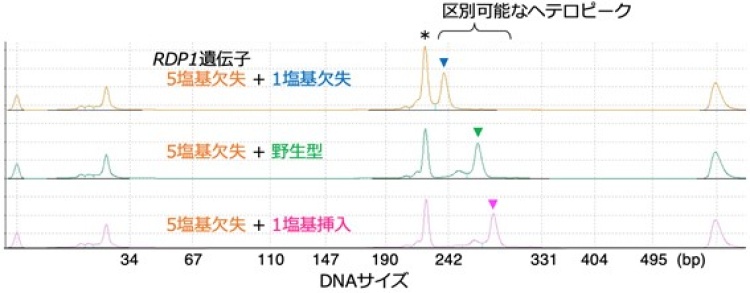 図2 プローブとして5塩基欠失配列を用いることで野生型配列と1塩基挿入/欠失配列を明確に判別することができた(矢頭)。星印はホモ二本鎖ピークを示す。
図2 プローブとして5塩基欠失配列を用いることで野生型配列と1塩基挿入/欠失配列を明確に判別することができた(矢頭)。星印はホモ二本鎖ピークを示す。
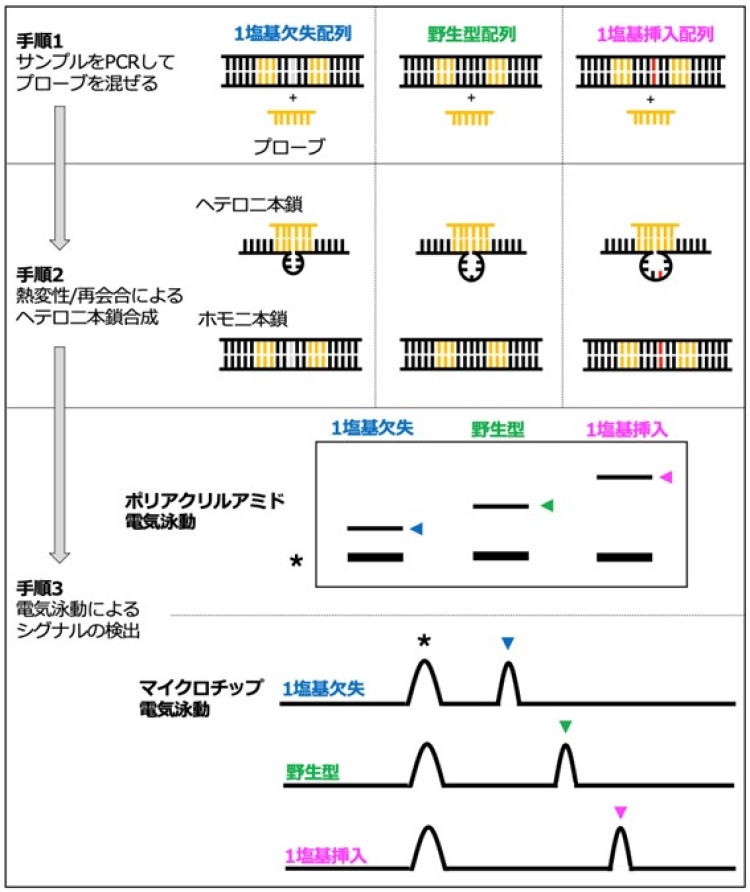 図3 PRIMAの標準プロトコル PCR, 熱変性/再会合, 電気泳動というシンプルな手順で簡便・迅速にDNAの1塩基差を判別することができる。電気泳動はポリアクリルアミド電気泳動もしくはマイクロチップ電気泳動のどちらの手法でも検出が可能。
図3 PRIMAの標準プロトコル PCR, 熱変性/再会合, 電気泳動というシンプルな手順で簡便・迅速にDNAの1塩基差を判別することができる。電気泳動はポリアクリルアミド電気泳動もしくはマイクロチップ電気泳動のどちらの手法でも検出が可能。
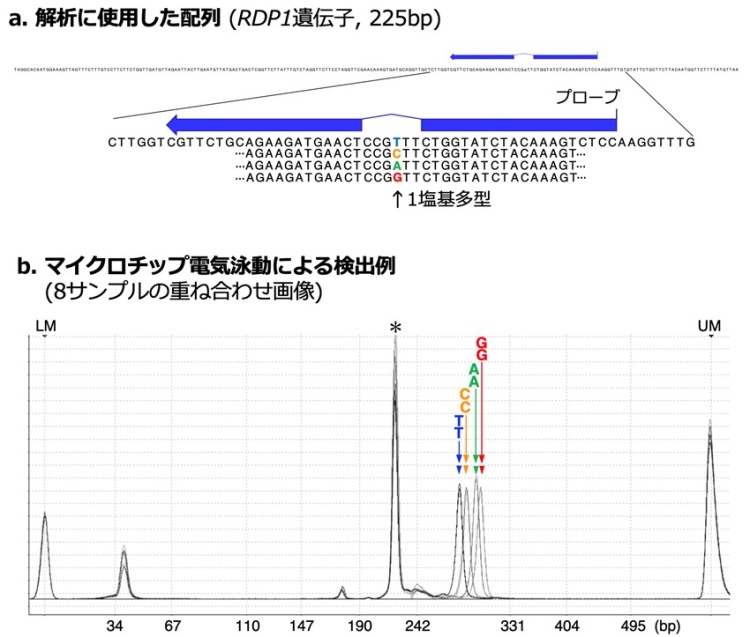 図4 PRIMAによる一塩基多型の判別 a. RDP1遺伝子の一ヶ所に一塩基多型を導入し、PRIMAにより判別が可能かどうか解析した。その結果、マイクロチップ電気泳動において、4種類の塩基を判別することが可能なヘテロピークが得られた(b)。
図4 PRIMAによる一塩基多型の判別 a. RDP1遺伝子の一ヶ所に一塩基多型を導入し、PRIMAにより判別が可能かどうか解析した。その結果、マイクロチップ電気泳動において、4種類の塩基を判別することが可能なヘテロピークが得られた(b)。
問い合わせ先
