2020.11.24
- プレスリリース
- 研究
遺伝情報の読み出しとDNA修復の両方に働く 生体超分子タンパク複合体の動的構造モデルを構築
~遺伝性難病の発症プロセスの解明や治療薬開発に期待~

横浜市立大学大学院生命医科学研究科の奥田昌彦特任准教授、浴本亨助教、栗田順一特任助教、池口満徳教授、西村善文特任教授(広島大学大学院統合生命科学研究科長兼任)は、NMR*1という分子構造を解析する分光器とスーパーコンピュータを駆使して、遺伝情報が読み出される転写反応やDNA修復反応に必須な転写因子TFIIH複合体の動的構造のモデル化に成功しました。また、癌抑制因子p53やDNA修復因子XPC等とTFIIHとのドッキング構造モデルを構築しました。この動的構造のモデルを用いることで、広範囲の疾患、色素性乾皮症やコケイン症等の遺伝性難病の発症原因とされる転写異常やDNA修復異常が起きる過程の解明、さらに疾患発症プロセスの解明や治療薬開発に役立つと考えられます。
本研究は、『Nucleic Acids Research』に掲載されました。(日本時間11月20日オンライン)
研究成果のポイント
|
研究の背景
ゲノム*2に記録されたヒトの遺伝情報は時空間的に正確に読み出されています。しかし、ゲノムDNAが保管されている細胞核は決して安全な場所とはいえず、紫外線、発癌物質等の体外からの攻撃に加え、代謝で生じた有害な副産物による体内からの攻撃も受け、絶えずゲノムDNAは傷ついています。このような危険の中で私たちが健康でいられるのは、長い進化の過程で獲得してきたDNAの修復機能のおかげです。遺伝情報が読み出される時やDNAが修復される際には数多くのタンパク質が関わり、それらが複雑に相互作用しながら反応を遂行して行きます。この相互作用がDNAの修復にとって決定的に重要である場合、たった一つの相互作用でも異常をきたすと反応効率低下や停止を引き起こし、病気、場合によっては死に至ってしまいます。従って、一つ一つの相互作用を詳しく調べることは、反応機構の解明、発症機構の解明に必須であり治療薬の開発につながります。
研究の内容
タンパク質間の相互作用を詳細に調べるにはまず、個々の立体構造を知る必要があります。本研究対象のTFIIHは十個の異なるタンパク質から構成される超分子複合体です。このTFIIH複合体のコアを形成するタンパク質の一つにp62があります。これまでのクライオ電子顕微鏡(クライオ電顕)による解析法では、コアの大部分の構造が見えましたが、p62のアミノ末端のPHドメインは外に露出してフレキシブルに動いているようで、その高い運動性のために全く構造を見ることができませんでした。このドメインは様々な転写因子や修復因子と特異的に相互作用してTFIIH複合体を適切な所へ先導する大切な役割を担っており、複合体中での振る舞いを理解することは構造の全貌を明らかにするだけでなく、標的タンパク質との相互作用を調べるためにも非常に重要です。
PHドメインのすぐ後に続くBSD1ドメインはクライオ電顕構造ではっきりと見えているため、PHドメインとBSD1ドメインを結ぶ短いリンカー領域が動いていることが想定され、この領域の運動性を詳しく知る必要があります。そのために私たちは、構造を決定することができ、且つ運動性も解析可能なNMR法を用いました。NMRは解析できる試料の分子量に上限があるため、高分子量のTFIIH複合体の解析には不向きです。しかし注目しているのはp62のPHドメインとリンカーであるため、クライオ電顕構造で見えていないPHドメインとリンカー、見えているBSD1ドメインを含んだ領域のみに対してNMRで解析しました。
NMRで得られた構造と運動性の情報を考慮し、クライオ電顕構造と組み合わせることにより、ヒトTFIIH複合体の動的な構造モデルを構築しました(図1上)。このうちの代表的な六個のモデルを初期構造としスーパーコンピュータにより分子動力学(MD)シミュレーションから精密化を行いました(図1下)。生体内では、タンパク質は水分子等の溶媒分子の中で機能を発現しています。MDを使うと、溶媒分子の中でタンパク質を動かすことができるため、TFIIH複合体構造モデルを生体内環境で精密化できます。
PHドメインのすぐ後に続くBSD1ドメインはクライオ電顕構造ではっきりと見えているため、PHドメインとBSD1ドメインを結ぶ短いリンカー領域が動いていることが想定され、この領域の運動性を詳しく知る必要があります。そのために私たちは、構造を決定することができ、且つ運動性も解析可能なNMR法を用いました。NMRは解析できる試料の分子量に上限があるため、高分子量のTFIIH複合体の解析には不向きです。しかし注目しているのはp62のPHドメインとリンカーであるため、クライオ電顕構造で見えていないPHドメインとリンカー、見えているBSD1ドメインを含んだ領域のみに対してNMRで解析しました。
NMRで得られた構造と運動性の情報を考慮し、クライオ電顕構造と組み合わせることにより、ヒトTFIIH複合体の動的な構造モデルを構築しました(図1上)。このうちの代表的な六個のモデルを初期構造としスーパーコンピュータにより分子動力学(MD)シミュレーションから精密化を行いました(図1下)。生体内では、タンパク質は水分子等の溶媒分子の中で機能を発現しています。MDを使うと、溶媒分子の中でタンパク質を動かすことができるため、TFIIH複合体構造モデルを生体内環境で精密化できます。
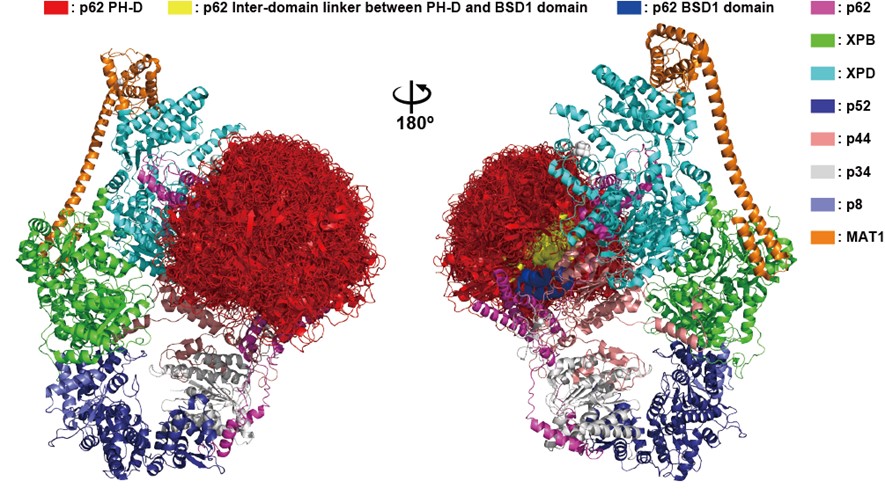
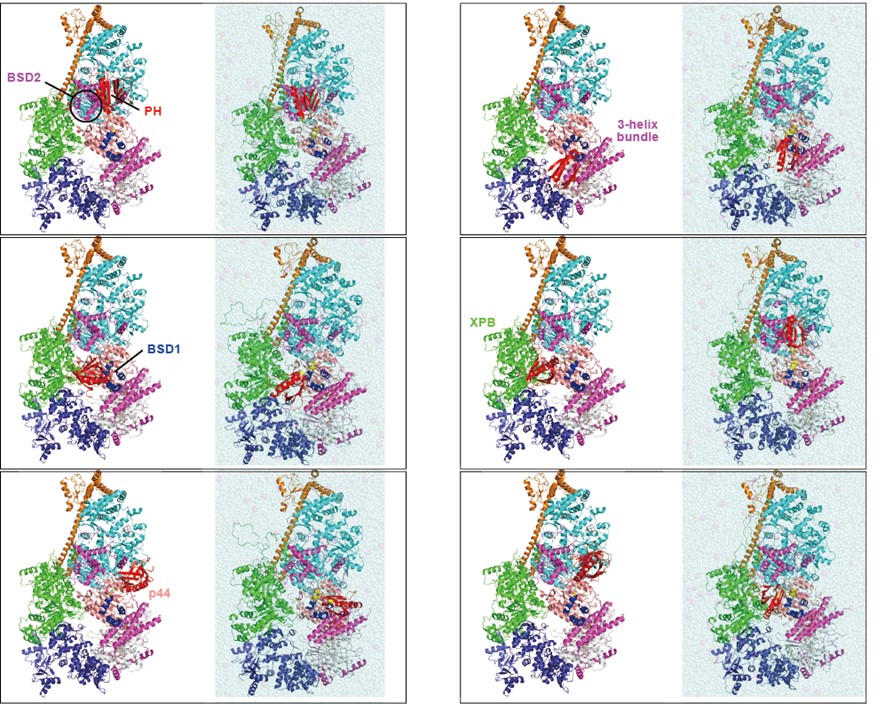
図1 ヒトTFIIH複合体の構造モデル
上:275個の構造の重ね合わせ。赤で示しているのがp62のPHドメイン。
下:代表的な六つの構造モデル。右は左の構造を初期構造としたMDシミュレーションから得られた構造。溶媒分子(シアン球)の中で構造モデルを精密化した。
上:275個の構造の重ね合わせ。赤で示しているのがp62のPHドメイン。
下:代表的な六つの構造モデル。右は左の構造を初期構造としたMDシミュレーションから得られた構造。溶媒分子(シアン球)の中で構造モデルを精密化した。
続いて、TFIIH複合体と基本転写因子TFIIEα、癌抑制因子p53、転写因子DP1、DNA修復因子のXPCとUVSSAとのドッキング構造を初期構造としスーパーコンピュータにより分子動力学(MD)シミュレーションから精密化を行いました(図2)。MDにより、溶媒分子を含む生体内環境で、TFIIHのPHドメインとドッキングした因子タンパク質の相互作用様式が精密化できました。
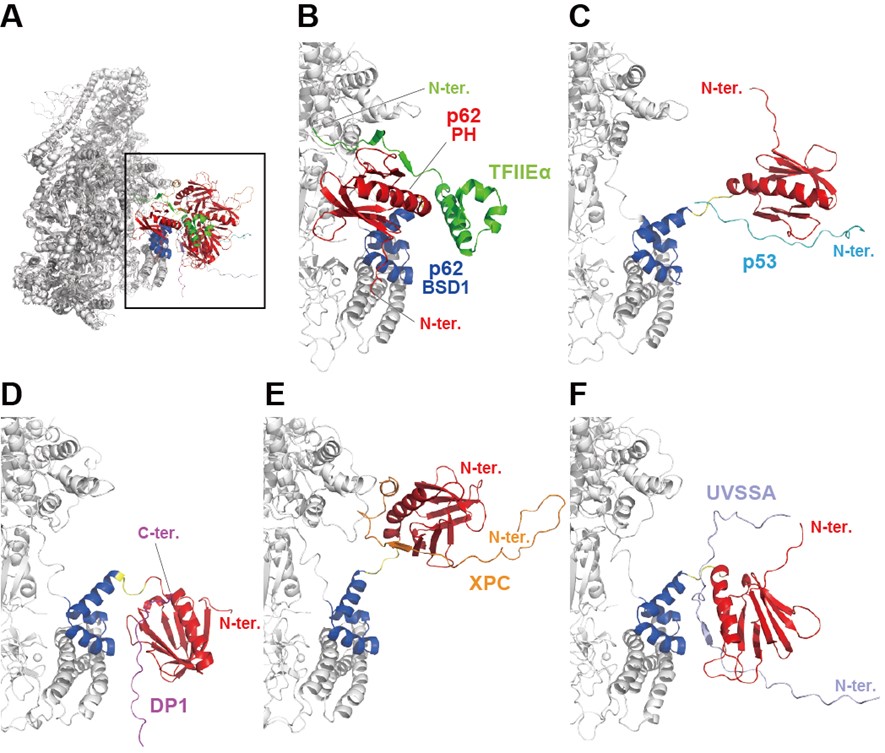
図2 ヒトTFIIH複合体と標的タンパク質とのMDシミュレーションから得られた複合体構造
以上の結果から、TFIIH複合体中でp62のPHドメインは自由に振る舞うものの、その後ろの五アミノ酸残基の短いリンカーにより特定の範囲内に限られることが分かりました。相互作用するタンパク質との複合体モデルは、転写やDNA修復反応が開始される初期段階の様子を表していると考えられます。
PHドメインの制限された運動は、相互作用する相手の選別に寄与しているのではないかと私たちは考えています。相手のタンパク質は自身では構造を形成しない領域(天然変性領域)を使い伸びた紐のような形でPHドメインと相互作用します。TFIIH複合体中でPHドメインがフレキシブルであるのは相手のタンパク質に認識されるのに優位になりますが、広範囲に動き過ぎると本命以外のタンパク質とも接触することで効率が低下してしまいます。その折衷案としてある程度運動範囲を制限することで、構造をもたない天然変性領域をもつ本命だけが有利に相互作用する仕組みになっているのではないかと推測されます。
以上の結果から、TFIIH複合体中でp62のPHドメインは自由に振る舞うものの、その後ろの五アミノ酸残基の短いリンカーにより特定の範囲内に限られることが分かりました。相互作用するタンパク質との複合体モデルは、転写やDNA修復反応が開始される初期段階の様子を表していると考えられます。
PHドメインの制限された運動は、相互作用する相手の選別に寄与しているのではないかと私たちは考えています。相手のタンパク質は自身では構造を形成しない領域(天然変性領域)を使い伸びた紐のような形でPHドメインと相互作用します。TFIIH複合体中でPHドメインがフレキシブルであるのは相手のタンパク質に認識されるのに優位になりますが、広範囲に動き過ぎると本命以外のタンパク質とも接触することで効率が低下してしまいます。その折衷案としてある程度運動範囲を制限することで、構造をもたない天然変性領域をもつ本命だけが有利に相互作用する仕組みになっているのではないかと推測されます。
今後の展開
現在多数のタンパク質から成る超分子複合体の構造がクライオ電顕で解析されるようになってきましたが、機能上重要な領域が動的に揺らいでいて見えない場合があります。そのような場合には本研究で示したようにクライオ電顕で見えていない領域をNMRで構造を解析し、そのドッキング構造をMDシミュレーションで精密化する方法が適用可能になります。今後は、転写反応や修復反応の一連の過程の中でTFIIH複合体を含めた様々なタンパク質との間で作られる相互作用を解明していくことを考えています。これにより、幅広い疾患の発症プロセスの解明や新たな治療薬開発に役立つことが期待されます。
用語説明
*1 NMR(Nuclear Magnetic Resonance):
強い磁場中で特定の原子核スピンの向きが揃えられた化合物やタンパク質等に対し、ラジオ波を照射して核磁気共鳴させた後、核スピンが元の安定な状態に戻る際に出す信号を観測して、原子の配置などを解析する装置。
*2 ゲノム:
遺伝情報の総体。遺伝子(gene)と染色体(chromosome)を組み合わせた用語で、生物をその生物たらしめるのに必須な最小限の染色体セットと定義されている。
強い磁場中で特定の原子核スピンの向きが揃えられた化合物やタンパク質等に対し、ラジオ波を照射して核磁気共鳴させた後、核スピンが元の安定な状態に戻る際に出す信号を観測して、原子の配置などを解析する装置。
*2 ゲノム:
遺伝情報の総体。遺伝子(gene)と染色体(chromosome)を組み合わせた用語で、生物をその生物たらしめるのに必須な最小限の染色体セットと定義されている。
論文情報
Structural and dynamical insights into the PH domain of p62 in human TFIIH
Masahiko Okuda, Toru Ekimoto, Jun-ichi Kurita, Mitsunori Ikeguchi, Yoshifumi Nishimura
Nucleic Acids Research https://doi.org/10.1093/nar/gkaa1045
※本研究は、国立研究開発法人日本医療研究開発機構(AMED)「創薬等ライフサイエンス研究支援基盤事業」、文部科学省「先端研究基盤共用促進事業(共用プラットフォーム形成支援プログラム) NMR共用プラットフォーム」の研究の一環で行われました。
Masahiko Okuda, Toru Ekimoto, Jun-ichi Kurita, Mitsunori Ikeguchi, Yoshifumi Nishimura
Nucleic Acids Research https://doi.org/10.1093/nar/gkaa1045
※本研究は、国立研究開発法人日本医療研究開発機構(AMED)「創薬等ライフサイエンス研究支援基盤事業」、文部科学省「先端研究基盤共用促進事業(共用プラットフォーム形成支援プログラム) NMR共用プラットフォーム」の研究の一環で行われました。
問い合わせ先
(取材対応窓口、資料請求など)
横浜市立大学 広報室
E-mail : koho@yokohama-cu.ac.jp