2020.05.18
- TOPICS
- 研究
脇役が助け合って主役を働かせる
~DNA修復に関わるタンパク質が助け合って働くメカニズムを解明~
横浜市立大学大学院生命医科学研究科 機能構造科学研究室の坂倉正義助教、高橋栄夫教授、東京工業大学の岩崎博史教授らの研究グループは、遺伝子の相同組み換えにおいて“主役”として働く酵素(リコンビナーゼRad51)を、“脇役”である補助タンパク質Sfr1-Swi5とRad55-Rad57が助け合いながら活性化するメカニズムを明らかにしました。
本研究のカギとなっている相同組み換えは、細胞が分裂する際に遺伝子を維持したり、遺伝子の多様性を生み出したりする反応であり、生命の根幹に関わる反応の一つです。その一方で、壊れた遺伝子DNAを元の状態に修復して、発癌を抑制するという機能も担っており、医学的に重要な反応でもあります。本研究成果を起点として、相同組み換えという理学的に興味深く、同時に医学的にも意義のある現象の分子メカニズムの解明が進むことが期待されます。
本研究成果は、バイオサイエンス分野の学術雑誌『eLife』に掲載されました。(日本時間 3月25日付)
本研究のカギとなっている相同組み換えは、細胞が分裂する際に遺伝子を維持したり、遺伝子の多様性を生み出したりする反応であり、生命の根幹に関わる反応の一つです。その一方で、壊れた遺伝子DNAを元の状態に修復して、発癌を抑制するという機能も担っており、医学的に重要な反応でもあります。本研究成果を起点として、相同組み換えという理学的に興味深く、同時に医学的にも意義のある現象の分子メカニズムの解明が進むことが期待されます。
本研究成果は、バイオサイエンス分野の学術雑誌『eLife』に掲載されました。(日本時間 3月25日付)
研究成果のポイント
|
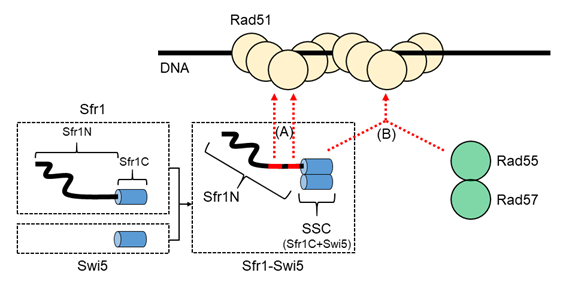
図1:補助タンパク質Sfr1-Swi5の一部分(Sfr1N)は、特定の立体構造を形成しない“天然変性タンパク質”である。 Sfr1-Swi5 は、Sfr1N上の2ヵ所の結合部位を介してRad51と結合し(A)、Rad51を活性化する。この結合が阻害された場合でも、Sfr1N領域があるとSfr1-Swi5は、Rad55-Rad57の補助によりRad51と結合し(B)、Rad51を活性化することができる。
研究の背景
生物の設計図である遺伝子DNAは、細胞分裂の過程において正確に複製され、娘細胞に分配される必要があります。ところがDNAは、紫外線、化学物質など、様々な外的要因により日常的に損傷を受けてしまうため、細胞は壊れたDNAを修復する様々なシステムを持っています。相同組み換えはこのようなシステムの一つであり、Rad51というタンパク質が中心となってDNAを修復します。
Rad51は単独ではDNAを修復することができず、補助タンパク質が結合することによって初めて機能を発揮します。補助タンパク質の一つであるSfr1-Swi5は、2つのタンパク質、Sfr1とSwi5、が強固に結合したタンパク質複合体です。Sfr1-Swi5は2つのパーツに分割でき、一つ目はSfr1のC末端領域(Sfr1C)とSwi5から成るパーツ(SSC)です(図1)。このパーツはRad51の活性化に必要不可欠な部分であることが明らかにされていますが、Rad51に結合する力が非常に弱く、Rad51を効率的に活性化することができません。一方、二つ目のパーツであるSfr1のN末端領域(Sfr1N)は、単独ではRad51を活性化することができませんが、Rad51と結合する力が強く、SSCとRad51との結合をアシストする役割を担っていると考えられます。
Sfr1-Swi5がRad51を活性化するメカニズムは未だに明らかになっていません。本研究ではこのメカニズムを解明する足掛かりとして、まずSfr1NがRad51に結合するメカニズムを解明しようと考え研究を行いました。
Rad51は単独ではDNAを修復することができず、補助タンパク質が結合することによって初めて機能を発揮します。補助タンパク質の一つであるSfr1-Swi5は、2つのタンパク質、Sfr1とSwi5、が強固に結合したタンパク質複合体です。Sfr1-Swi5は2つのパーツに分割でき、一つ目はSfr1のC末端領域(Sfr1C)とSwi5から成るパーツ(SSC)です(図1)。このパーツはRad51の活性化に必要不可欠な部分であることが明らかにされていますが、Rad51に結合する力が非常に弱く、Rad51を効率的に活性化することができません。一方、二つ目のパーツであるSfr1のN末端領域(Sfr1N)は、単独ではRad51を活性化することができませんが、Rad51と結合する力が強く、SSCとRad51との結合をアシストする役割を担っていると考えられます。
Sfr1-Swi5がRad51を活性化するメカニズムは未だに明らかになっていません。本研究ではこのメカニズムを解明する足掛かりとして、まずSfr1NがRad51に結合するメカニズムを解明しようと考え研究を行いました。
研究の内容
先行研究において、Sfr1Nはそのアミノ酸配列から天然変性タンパク質*1であることが予想されていました。天然変性タンパク質は特定の形を持っていないタンパク質であり、X線結晶構造解析など分子の形を見る手法では解析することができません。そこで本研究では、天然変性タンパク質を解析することが可能な唯一の手法である核磁気共鳴(NMR)*2装置を用いてSfr1Nの性状を明らかにしようと試みました。NMRを用いてSfr1Nを構成するアミノ酸残基が隣のアミノ酸残基と成す角度や、各アミノ酸残基の動きやすさを解析した結果、Sfr1Nが予想通り天然変性タンパク質であり、溶液中において柔らかい紐のような状態で存在していることが明らかとなりました。
次に、Sfr1N上のRad51結合部位を明らかにするために、Sfr1NにRad51を加え、NMR信号の変化を解析しました(図2A)。この結果、Sfr1N上には我々がSite 1、Site 2と名付けた2か所のRad51結合部位が存在することが明らかとなりました(図2B)。Site 1、Site 2上のアミノ酸残基を他のアミノ酸残基(Ala残基)に置き換えたSfr1-Swi5の変異体を用いてRad51のDNA相同組換え反応を行うと、野生型(変異無し)> Site 1変異体 ≈ Site 2変異体 > Site 1&2二重変異体 の順に活性が低下したことから、Site 1とSite 2が協同的に働いていることが分かりました。
次に、Sfr1N上のRad51結合部位を明らかにするために、Sfr1NにRad51を加え、NMR信号の変化を解析しました(図2A)。この結果、Sfr1N上には我々がSite 1、Site 2と名付けた2か所のRad51結合部位が存在することが明らかとなりました(図2B)。Site 1、Site 2上のアミノ酸残基を他のアミノ酸残基(Ala残基)に置き換えたSfr1-Swi5の変異体を用いてRad51のDNA相同組換え反応を行うと、野生型(変異無し)> Site 1変異体 ≈ Site 2変異体 > Site 1&2二重変異体 の順に活性が低下したことから、Site 1とSite 2が協同的に働いていることが分かりました。
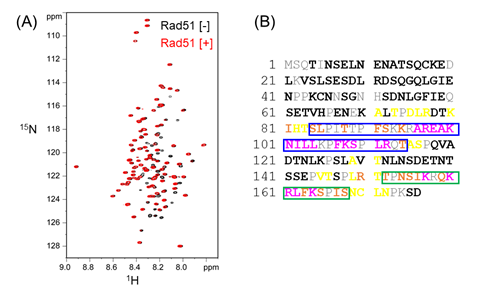
図2:(A) Rad51非存在下(黒)、Rad51存在下(赤)において測定したSfr1NのNMRスペクトルの重ね合わせ。スペクトル中の一つのシグナルが一つのアミノ酸残基に対応している。
(B) Rad51添加に伴いNMRシグナルの強度が40-60%(黄)、60-80%(オレンジ)、80%以上(マジェンタ)低下した残基をSfr1Nのアミノ酸配列上にマッピングした結果。Site 1 (Ser84~Thr114、青い四角)およびSite 2 (Thr152~Ser168、緑の四角)の二つの領域がRad51と相互作用することが示された。
(B) Rad51添加に伴いNMRシグナルの強度が40-60%(黄)、60-80%(オレンジ)、80%以上(マジェンタ)低下した残基をSfr1Nのアミノ酸配列上にマッピングした結果。Site 1 (Ser84~Thr114、青い四角)およびSite 2 (Thr152~Ser168、緑の四角)の二つの領域がRad51と相互作用することが示された。
次に、Site 1、Site 2を変異させた酵母変異株に紫外線を照射してDNAに損傷を与え、その後細胞が生存することができるかどうかを調べました。すると、Site 1、Site 2の変異によりRad51の相同組み換え反応活性が低下し、DNA損傷を修復できずに細胞が死ぬという予想に反して、Site 1・Site 2変異株は生育可能であり、一見Site 1・Site 2を介したSfr1-Swi5とRad51の結合が、DNA修復において不必要であるかのような結果が得られました。
酵母にはSfr1-Swi5とは別の補助因子であるRad55-Rad57が発現しており、このRad55-Rad57がSfr1-Swi5の代役として機能した可能性が考えられました。そこで、Rad55またはRad57を欠損させた酵母株に、Site 1・Site 2変異を導入したところ、予想通り、酵母細胞はDNAを修復できず、死んでしまいました。
また、DNA損傷を受けたSite 1・Site 2変異株の生存率が、Sfr1全体を欠損した変異株より高かったことから、Site 1・Site 2変異株では、Rad55-Rad57がRad51を直接的に活性化しているだけではなく、Sfr1-Swi5変異体(Site 1・Site 2変異体)を介したRad51の活性化もサポートしていると考えられました。そこで、免疫沈降法によりRad55-Rad57とSfr1-Swi5の相互作用の有無を解析したところ、両者の直接的な結合が見られました。これらの結果から、Rad55-Rad57が物理的にSfr1-Swi5とRad51の結合を橋渡しする役割を担うことが明らかとなりました。
これまでSfr1-Swi5とRad55-Rad57は、互いに無関係に働くと考えられてきましたが、本研究の結果から、細胞内においてRad51の補助因子が助け合って働くメカニズムが存在することが初めて示されました。
酵母にはSfr1-Swi5とは別の補助因子であるRad55-Rad57が発現しており、このRad55-Rad57がSfr1-Swi5の代役として機能した可能性が考えられました。そこで、Rad55またはRad57を欠損させた酵母株に、Site 1・Site 2変異を導入したところ、予想通り、酵母細胞はDNAを修復できず、死んでしまいました。
また、DNA損傷を受けたSite 1・Site 2変異株の生存率が、Sfr1全体を欠損した変異株より高かったことから、Site 1・Site 2変異株では、Rad55-Rad57がRad51を直接的に活性化しているだけではなく、Sfr1-Swi5変異体(Site 1・Site 2変異体)を介したRad51の活性化もサポートしていると考えられました。そこで、免疫沈降法によりRad55-Rad57とSfr1-Swi5の相互作用の有無を解析したところ、両者の直接的な結合が見られました。これらの結果から、Rad55-Rad57が物理的にSfr1-Swi5とRad51の結合を橋渡しする役割を担うことが明らかとなりました。
これまでSfr1-Swi5とRad55-Rad57は、互いに無関係に働くと考えられてきましたが、本研究の結果から、細胞内においてRad51の補助因子が助け合って働くメカニズムが存在することが初めて示されました。
今後の展開
本研究によりSfr1N上のRad51結合部位が決定されましたが、Sfr1-Swi5がRad51を活性化する分子メカニズムの大部分は依然として謎のまま残されています。今回明らかにしたSite 1・Site 2を起点とすることにより、Rad51上のSfr1N結合部位を探索するなど、相同組換えの分子メカニズムを解明するための新たな研究の進展が期待されます。
用語説明
*1 天然変性タンパク質:一般的なタンパク質は、そのアミノ酸配列に応じて独特の立体構造を形成し、これによって初めて機能を発揮する。これに対して天然変性タンパク質は、一定の構造を形成せず、柔らかい紐のような状態で存在しつつ、機能を発揮することができる。
*2 核磁気共鳴(法)(NMR):磁場中で原子核の共鳴信号を検出する分光学手法。原子ごとに共鳴信号の周波数がわずかに異なるため、原子ごとに個別の情報を取り出すことができる。天然変性タンパク質のような構造的な特徴が少ない生体分子を解析する場合には、強力な磁場を発生させる大型NMR装置が有効となる。横浜市立大学は、日本で最も高い磁場を発生させる950 MHz NMR装置など、複数の高磁場NMR装置を保有しており、本研究ではこれらの装置を駆使して研究を行った。
*2 核磁気共鳴(法)(NMR):磁場中で原子核の共鳴信号を検出する分光学手法。原子ごとに共鳴信号の周波数がわずかに異なるため、原子ごとに個別の情報を取り出すことができる。天然変性タンパク質のような構造的な特徴が少ない生体分子を解析する場合には、強力な磁場を発生させる大型NMR装置が有効となる。横浜市立大学は、日本で最も高い磁場を発生させる950 MHz NMR装置など、複数の高磁場NMR装置を保有しており、本研究ではこれらの装置を駆使して研究を行った。
掲載論文
Cooperative interactions facilitate stimulation of Rad51 by the Swi5-Sfr1 auxiliary factor complex
Bilge Argunhan*1, Masayoshi Sakakura*1, Negar Afshar, Misato Kurihara, Kentaro Ito, Takahisa Maki, Shuji Kanamaru, Yasuto Murayama, Hideo Tsubouchi, Masayuki Takahashi, Hideo Takahashi*2, Hiroshi Iwasaki*2
*1Co-first authors
*2Corresponding authors
eLife(2020), https://doi.org/10.7554/eLife.52566
※本研究は、JSPS科学研究費(JP18H04626、JP18H05426)、横浜市立大学基礎研究費などの助成を受けて行われました。
問い合わせ先
(内容に関するお問い合わせ)
大学院生命医科学研究科 機能構造科学研究室
教授 高橋栄夫
TEL : 045-508-7214
E-mail : hidtak@yokohama-cu.ac.jp