2019.01.15
- プレスリリース
- 研究
UHRF1とLIG1の複合体構造解析
~阻害剤開発の基盤となる相互作用部位を同定~
『Structure』に掲載(1月10日付オンライン)
横浜市立大学 大学院生命医科学研究科 構造生物学研究室 有田恭平准教授、郡聡実 (博士前期課程)、Centre national de la recherche scientifique (CNRS;フランス国立科学研究センター) Pierre-Antoine Defossezグループディレクターの国際共同研究グループは、DNAリガーゼLIG1※1とUHRF1※2の複合体の立体構造を決定し、DNAメチル化※3の継承機構の基盤を解明しました。
|
研究成果のポイント
|
研究の背景
ヒトの体を構成するおよそ200種類以上の細胞は、同じ遺伝子のセットを持つにもかかわらず形や機能が異なります。これは、各細胞で使われる遺伝子が異なるためです。その使われ方を決める因子の一つがDNAメチル化です。細胞では分裂の前にDNAが正確に複製され親細胞から娘細胞に受け継がれますが、DNAメチル化も同様です。この「DNA維持メチル化」とよばれるプロセスが正常でないと、細胞のがん化など様々な不具合が生じるので、DNAメチル化パターンの厳密な制御と継承が必要です。
DNA維持メチル化にはUHRF1とDNA維持メチル化酵素DNMT1の2つのタンパク質が必須の役割をします。UHRF1はDNA複製後に生じた片鎖メチル化DNAを認識し、ここにDNMT1を呼び込む働きをします。有田准教授らは、この呼び込みはリジン126がメチル化されたDNAリガーゼ1 (LIG1K126me3 )によることを2017年8月に報告しました (Molecular Cell 67, 550-565, August 17, 2017)。
UHRF1には5つの機能ドメインがあり、そのうち特徴的な「ペプチド結合溝」をもつTTDドメイン※4 (以下UHRF1 TTD) がLIG1K126me3と結合します。この溝は他にも、9番目のリジンがメチル化されたヒストンH3 ※5(H3K9me3) や、UHRF1の分子内にあるリンカー領域が結合しますが、興味深いことに、LIG1K126me3のUHRF1 TTDへの結合は他の結合因子に比べ180倍以上も強いことが知られています。このUHRF1とLIG1の高親和性の結合が、UHRF1を正確に複製サイトに呼び込むために必要と考えられますが、その結合をもたらす仕組みは不明でした。また、複数の機能ドメインから成るUHRF1は、基質の結合が高次構造を変化させその機能を制御していることが考えられますが、UHRF1の高次構造の変化はよくわかっていませんでした。そこで本研究グループは、UHRF1とLIG1の複合体の構造と、UHRF1の高次構造の変化の解析を行うことにより、DNA維持メチル化の機構解明に取り組みました。
DNA維持メチル化にはUHRF1とDNA維持メチル化酵素DNMT1の2つのタンパク質が必須の役割をします。UHRF1はDNA複製後に生じた片鎖メチル化DNAを認識し、ここにDNMT1を呼び込む働きをします。有田准教授らは、この呼び込みはリジン126がメチル化されたDNAリガーゼ1 (LIG1K126me3 )によることを2017年8月に報告しました (Molecular Cell 67, 550-565, August 17, 2017)。
UHRF1には5つの機能ドメインがあり、そのうち特徴的な「ペプチド結合溝」をもつTTDドメイン※4 (以下UHRF1 TTD) がLIG1K126me3と結合します。この溝は他にも、9番目のリジンがメチル化されたヒストンH3 ※5(H3K9me3) や、UHRF1の分子内にあるリンカー領域が結合しますが、興味深いことに、LIG1K126me3のUHRF1 TTDへの結合は他の結合因子に比べ180倍以上も強いことが知られています。このUHRF1とLIG1の高親和性の結合が、UHRF1を正確に複製サイトに呼び込むために必要と考えられますが、その結合をもたらす仕組みは不明でした。また、複数の機能ドメインから成るUHRF1は、基質の結合が高次構造を変化させその機能を制御していることが考えられますが、UHRF1の高次構造の変化はよくわかっていませんでした。そこで本研究グループは、UHRF1とLIG1の複合体の構造と、UHRF1の高次構造の変化の解析を行うことにより、DNA維持メチル化の機構解明に取り組みました。
研究の内容
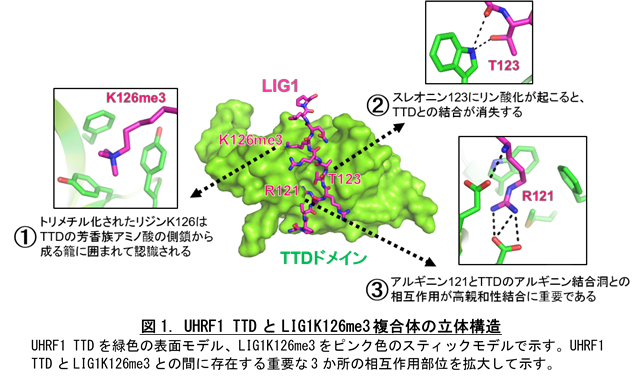
まず、UHRF1 TTDとLIG1K126me3ペプチド複合体の立体構造を大型放射光施設Photon Factory (PF) のBL-17Aを用いてX線結晶構造解析法で決定しました。その結果、LIG1K126me3はUHRF1 TTDのペプチド結合溝に結合していました。そして、そのTTDとLIG1K126me3との間には重要な相互作用部位が3か所あることがわかりました (図1)。
1つめはLIG1のトリメチル化されたリジン126とTTDの芳香族アミノ酸の側鎖から成る籠との相互作用です。2つめは、LIG1のスレオニン123とUHRF1 TTDとの間の水素結合です。このLIG1のスレオニン123にリン酸化が起こると、TTDとの結合が消失することがわかり、LIG1のリン酸化がUHRF1との結合を制御するスイッチの役割をしていることを明らかにしました。3つめは、LIG1のアルギニン121とTTDの「アルギニン結合洞」と名付けたポケットとの間の相互作用です。別のUHRF1結合タンパク質であるヒストンH3と比較すると、LIG1のアルギニン121に相当するアミノ酸はヒストンH3ではリジン4でした。このヒストンH3のリジン4をアルギニン残基に変異させたところ、UHRF1 TTDへの親和性がLIG1とほぼ同じになり、LIG1のアルギニン121とUHRF1 TTDのアルギニン結合洞との相互作用が高親和性を決める重要なポイントであることがわかりました。そして、立体構造解析から明らかにしたUHRF1 TTDとLIG1K126me3の相互作用に重要と考えられるアミノ酸残基に部位特異的な変異を導入すると、実際に結合が大幅に減少することを生化学と哺乳類細胞を用いた相互作用解析 (CNRS, Pierre-Antoine Defossez氏) により明らかにしました。
1つめはLIG1のトリメチル化されたリジン126とTTDの芳香族アミノ酸の側鎖から成る籠との相互作用です。2つめは、LIG1のスレオニン123とUHRF1 TTDとの間の水素結合です。このLIG1のスレオニン123にリン酸化が起こると、TTDとの結合が消失することがわかり、LIG1のリン酸化がUHRF1との結合を制御するスイッチの役割をしていることを明らかにしました。3つめは、LIG1のアルギニン121とTTDの「アルギニン結合洞」と名付けたポケットとの間の相互作用です。別のUHRF1結合タンパク質であるヒストンH3と比較すると、LIG1のアルギニン121に相当するアミノ酸はヒストンH3ではリジン4でした。このヒストンH3のリジン4をアルギニン残基に変異させたところ、UHRF1 TTDへの親和性がLIG1とほぼ同じになり、LIG1のアルギニン121とUHRF1 TTDのアルギニン結合洞との相互作用が高親和性を決める重要なポイントであることがわかりました。そして、立体構造解析から明らかにしたUHRF1 TTDとLIG1K126me3の相互作用に重要と考えられるアミノ酸残基に部位特異的な変異を導入すると、実際に結合が大幅に減少することを生化学と哺乳類細胞を用いた相互作用解析 (CNRS, Pierre-Antoine Defossez氏) により明らかにしました。
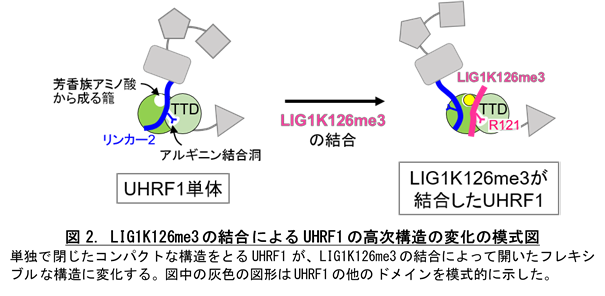
さらに、LIG1K126me3の結合によるUHRF1の高次構造の変化をX線溶液散乱※6 (PF BL-10C) や高速原子間力顕微鏡※7 (金沢大学バイオAFM先端研究センター) を用いて解析しました。UHRF1単体では、TTDのペプチド結合溝にはUHRF1分子内のリンカー2が結合しており、閉じたコンパクトな構造をとります (図2左)。しかし、LIG1K126me3がTTDのペプチド結合溝に結合すると、リンカー2はペプチド結合溝から追い出され、その結果UHRF1は開いたフレキシブルな構造に変わることが本研究で初めて明らかになり、この高次構造の変化がUHRF1の機能制御に重要であることが示唆されました (図2右)。
今後の展開
本研究成果は、DNA維持メチル化に関わるUHRF1がどのようにして複製因子LIG1により複製の起きている場所に連れてこられるかを構造生物学的な観点から初めて解明しました。DNA維持メチル化と複製の連携については今まさに研究が始まったばかりであり、本研究はこの研究分野の先駆的な成果となります。
DNA維持メチル化の異常は、がんや精神疾患などの様々な病気との関連が報告されています。UHRF1はDNAメチル化を維持する重要な因子であるとともに、様々ながん細胞で高発現して異常な増殖に関与することから、薬剤の標的分子になると考えられています。本研究のX線結晶構造解析から、LIG1のアルギニン121がUHRF1 TTDの「アルギニン結合洞」に入ることが高親和性結合を実現する重要な相互作用であることがわかりました。つまり、TTDの「アルギニン結合洞」に結合する化合物はUHRF1のTTDの機能を強力に阻害し、がん細胞で過剰に発現したUHRF1の働きを抑えられると考えられます。今回の研究成果はDNA維持メチル化の生物学的な意義を明らかにするとともに、この分野の応用への寄与が期待されます。
DNA維持メチル化の異常は、がんや精神疾患などの様々な病気との関連が報告されています。UHRF1はDNAメチル化を維持する重要な因子であるとともに、様々ながん細胞で高発現して異常な増殖に関与することから、薬剤の標的分子になると考えられています。本研究のX線結晶構造解析から、LIG1のアルギニン121がUHRF1 TTDの「アルギニン結合洞」に入ることが高親和性結合を実現する重要な相互作用であることがわかりました。つまり、TTDの「アルギニン結合洞」に結合する化合物はUHRF1のTTDの機能を強力に阻害し、がん細胞で過剰に発現したUHRF1の働きを抑えられると考えられます。今回の研究成果はDNA維持メチル化の生物学的な意義を明らかにするとともに、この分野の応用への寄与が期待されます。
用語解説
*1 LIG1
DNA複製中のラギング鎖で生じた岡崎フラグメントを連結する酵素。複製が起こっている場所に局在しており、UHRF1を複製されている場所に呼び込む働きも持つ。
*2 UHRF1
DNAメチル化維持に必須の役割をするタンパク質。片鎖メチル化DNAに結合したり、9番目のリジンがメチル化されたヒストンH3に結合したり、ヒストンH3をユビキチン化するなど様々な機能を発揮することで、DNAメチル化パターンの複製を誘導する。がん細胞で過剰発現しており、異常な細胞増殖に関与する。
*3 DNAメチル化
DNAに含まれるシトシン塩基にメチル基(-CH3)が付加される反応。ヒトではCG配列中のシトシン塩基のみがメチル化される。DNAメチル化により、遺伝子の発現が抑制されると考えられている。生物の体(多細胞の形質)を形成するために必須である。
*5 ヒストンH3
DNA複製中のラギング鎖で生じた岡崎フラグメントを連結する酵素。複製が起こっている場所に局在しており、UHRF1を複製されている場所に呼び込む働きも持つ。
*2 UHRF1
DNAメチル化維持に必須の役割をするタンパク質。片鎖メチル化DNAに結合したり、9番目のリジンがメチル化されたヒストンH3に結合したり、ヒストンH3をユビキチン化するなど様々な機能を発揮することで、DNAメチル化パターンの複製を誘導する。がん細胞で過剰発現しており、異常な細胞増殖に関与する。
*3 DNAメチル化
DNAに含まれるシトシン塩基にメチル基(-CH3)が付加される反応。ヒトではCG配列中のシトシン塩基のみがメチル化される。DNAメチル化により、遺伝子の発現が抑制されると考えられている。生物の体(多細胞の形質)を形成するために必須である。
*4 TTDドメイン(UHRF1 TTD)
UHRF1が有する機能ドメイン。Tudorドメインがタンデムに2つ並んだ構造をとり、その間に形成されるペプチド結合溝で、ヘテロクロマチン形成の指標であるH3K9me3や、UHRF1分子内のリンカーと相互作用する。
*5 ヒストンH3
ゲノムDNAはヒストンH2A, H2B, H3, H4から成るヒストン8量体に巻き付いてヌクレオソームという構造体をとり、コンパクトに折りたたまれて核の中に収納される。ヒストンH3の9番目のリジン残基のトリメチル化はヌクレオソームの高度な凝縮に関与している。
*6 X線溶液散乱
*6 X線溶液散乱
溶液中のタンパク質にX線を照射し、得られた散乱強度データからタンパク質の構造情報を得る手法。低分解能であるが溶液中のタンパク質の平均構造がわかり、タンパク質の構造変化を解析できる。
*7 高速原子間力顕微鏡
先端を尖らせた探針を用いて、タンパク質の表面をなぞるように動かす走査型顕微鏡。タンパク質一分子の構造と動態をリアルタイムで観察することができる。
※本研究は、『Structure』に掲載されました。(1月10日付オンライン)
問い合わせ先
(研究内容に関するお問合せ)
大学院生命医科学研究科 構造生物学
准教授 有田 恭平
TEL:045-508-7227
E-mail: aritak@yokohama-cu.ac.jp
(取材対応窓口、資料請求など)
研究企画・産学連携推進課長 渡邊 誠
TEL:045-787-2510
E-mail: kenkyupr@yokohama-cu.ac.jp
大学院生命医科学研究科 構造生物学
准教授 有田 恭平
TEL:045-508-7227
E-mail: aritak@yokohama-cu.ac.jp
(取材対応窓口、資料請求など)
研究企画・産学連携推進課長 渡邊 誠
TEL:045-787-2510
E-mail: kenkyupr@yokohama-cu.ac.jp