- プレスリリース
- 研究
ポリユビキチンによる柔軟でダイナミックな分子認識機構を解明
~科学雑誌『Scientific Reports』 に掲載~
この研究の成果は、横浜市立大学鶴見キャンパスのスーパーコンピュータを用いた大規模な計算機シミュレーションによって得られました。ユビキチンが2つつながったジユビキチンとそれが特異的に認識するタンパク質であるユビキチン結合ドメイン(UBD)との複合体は、通常のタンパク質複合体に見られる1つの安定構造とは異なり、極めて多様な結合様式の集合体として存在していること(ダイナミックな分子認識機構)を見出しました。さらに、様々な基質タンパク質の特異性を決定する機構として知られているポリユビキチンの重合のしかた(リンケージ)が、どのようにしてこのようにダイナミックな分子認識機構の中で基質分子を見分けているのかを明らかにしました。
|
研究成果のポイント
〇スパコンを用いた大規模な計算機シミュレーションにより、ジユビキチンとユビキチン結合ドメインとの複合体の分子認識機構を明らかにした。 〇ポリユビキチン鎖の多様なリンケージにより、様々なユビキチン結合ドメインを特異的に認識する分子機構を見出した。 |
研究の背景
76個のアミノ酸からなる小さなタンパク質ユビキチンは、重合してポリユビキチンとなり、タンパク質の修飾や、タンパク質との相互作用を通じて、タンパク質分解やDNA修復、シグナル伝達など多彩な細胞機能を担っています。ポリユビキチン鎖は、ユビキチンのC末端残基が他のユビキチンの7つのLys残基のひとつ、もしくはN末端残基の8通りのいずれかと共有結合(リンケージ)することで、多様な重合構造を形成します。異なるリンケージを持つポリユビキチンは、それぞれのリンケージの立体構造に対応した基質分子(ユビキチン結合ドメイン: UBD)と特異的に結合することで、異なった機能を発現します。ポリユビキチン鎖はリンケージの組み合わせによって多様なUBDとの特異的相互作用を実現すると考えられますが、その仕組みを理解するには、ポリユビキチン鎖の最小単位である2つのユビキチンが結合したタンパク質(ジユビキチン)とUBDとのタンパク質間相互作用のあり方をシミュレーションから知ることが鍵になると考え、本研究を行いました。
研究の概要と成果
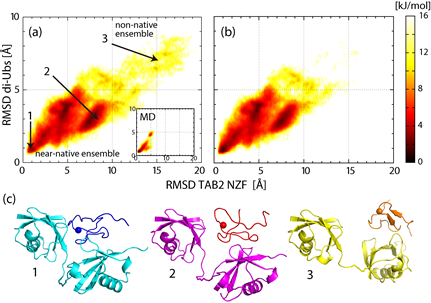
詳細を図2に示します。図2(a)は、構造分布を表現した図で、左下が実験的に観察された結晶構造にあたり、右上になるほど、ふたつのユビキチン間およびユビキチンとNZF間の配置がずれていることを示します。ふたつのユビキチン分子の配置については10Å近く、NZFドメインの配置は20Å近くまでずれた構造があり、それらが連続的につながった幅広い構造分布を持っていることが分かります。このように広い構造分布を持つにもかかわらず、完全に解離してしまうことはなく、結晶構造を中心に大きくゆらいでいました。一つの立体配置をとっているタンパク質複合体の結晶構造とはまったく異なる、ダイナミックな分子認識機構がはじめてシミュレーションによって明らかにされました。同様にMSES法を用いたシミュレーション研究で明らかとなった、強く結合する酵素/阻害タンパク質複合体(Barnase/Barstar複合体)では、一つの安定な構造が支配的なファネル型の構造分布をとることが明らかとなっており(参考文献2)、ジユビキチン複合体ではそれとはまったく異なる極めてダイナミックなものであることが分かりました。
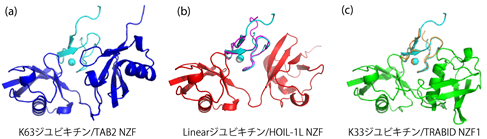
ジユビキチンのリンケージに対応した基質の特異性がどのようにして現れるかは、同様にスーパーコンピュータによるシミュレーションによって調べました。まず、TAB2 NZFが異なったリンケージのジユビキチンに結合したらどうなるかを、図1に示した相同なNZFドメインの複合体に基づいた非天然の複合体モデル構造(Linearジユビキチン/ TAB2 NZFとK33ジユビキチン/ TAB2 NZF)のシミュレーションによって検証しました。その結果、ふたつの非天然複合体モデルは不安定であり、容易に解離してしまうことが分かりました。これは、これらのリンケージのジユビキチンの表面にはTAB2 NZFを正しく結合するために必要な相互作用をする残基がないことが原因でした。天然のLinearジユビキチン/HOIL-1L NZFとK33ジユビキチン/TRABID NZF1が、K63ジユビキチン/TAB2 NZFと同等の安定性を示すことをシミュレーションで確認したこととあわせて、ダイナミックな分子認識においても、1つの安定な立体配置をとる複合体と同様に複合体界面での相補的な相互作用が重要な役割を果たしていることが示されました。
上記は、異なったリンケージに現れる異なった相互作用面を使うモデルによる安定性の検証でしたが、さらに相互作用面をK63のものに維持したままリンケージのみをつなぎかえたモデルの安定性についてもシミュレーションで検証しました(linear、K6、K11、K48ジユビキチン)。その結果、全てが不安定化したことが確認されました。不安定化は、つなぎかえでリンカーが伸びきったため縮もうとした(linear、K11)、つなぎかえでリンカーに立体障害が生じた(K48)という原因によって起こっていました。一方、K6ジユビキチンでは、リンカーにつなぎかえで天然のK63よりも長さに余裕ができ、立体障害も起こらないにもかかわらず不安定化したのは、K63におけるリンカーの適切な長さが2つのユビキチン間の相互配置を最適に維持していることを示していると考えられました。
議論と今後の展開
これからも、スーパーコンピュータによるシミュレーションを用いて、このようにダイナミックな分子機能発現過程をさらに多くの生物学の問題について研究していく予定です。
用語説明
*2 MSES法:シミュレーションによってタンパク質の幅広い構造分布を計算する方法。アミノ酸をひとつの質点として簡単化した粗視化モデルと溶媒分子とタンパク質からなる全原子モデルを連成した方法で、大きなタンパク質の広い構造分布の同定に適している。
参考文献
2. Kei Moritsugu, Tohru Terada and Akinori Kidera, “Energy Landscape of All-Atom Protein-Protein Interactions Revealed by Multiscale Enhanced Sampling,” PLoS Computational Biology (2014) 10: e1003901.
論文著者、並びにタイトルなど
Dynamic recognition and linkage specificity in K63 di-ubiquitin and TAB2 NZF domain complex
Kei Moritsugu1, Hafumi Nishi1,2, Keiichi Inariyama1, Masanori Kobayashi1 & Akinori Kidera1
Scientific Reports.2018 Nov 7;8(1):16478. doi: 10.1038/s41598-018-34605-2.
1 横浜市立大学大学院生命医科学研究科
2 現所属:東北大学大学院情報科学研究科
大学院生命医科学研究科 教授 木寺詔紀
TEL:045-508-7231
E-mail:kidera@yokohama-cu.ac.jp
(取材対応窓口、資料請求など)
研究企画・産学連携推進課長 渡邊 誠
TEL:045-787-2510
E-Mail:kenkyupr@yokohama-cu.ac.jp