2016.03.04
- プレスリリース
- 研究
鶴見キャンパスの研究グループが、遺伝子発現を抑えるヘテロクロマチン構造の形成機構を解明
~がんの発生やiPS細胞産生の原理解明に向けて~
平成28年3月3日
研究推進課
研究推進課
Nature Publishing Group『Scientific Reports』(平成28年3月3日オンライン版)に掲載
横浜市立大学大学院生命医科学研究科 下條秀朗元特任助教、木寺詔紀教授、佐藤衛教授、西村善文学長補佐は、名古屋市立大学 中山潤一教授らとの共同研究で、遺伝子の発現が抑えられるヘテロクロマチン構造形成に関与するタンパク質の結合機構を解明しました。
タンパク質の紐様構造を横浜市大鶴見キャンパスに設置した世界最高級感度を誇るNMR分光器、スーパーコンピュータ、実験室内小型X線小角散乱装置を用いて解析しました。今回の解析により、タンパク質から伸びた紐様構造のタンパク質同士が互いに結合して、そのタンパク質の結合を強くしていることを世界で初めて明らかにしました。特にクロマチン構造に関連するタンパク質においては、紐様構造のタンパク質同士の相互作用が重要であることが分かり、タンパク質は球状構造を取らない紐様構造でも機能することを解明しました。
今回の成果は、教科書等で確立されている、タンパク質が機能するときは球状構造を取り紐状構造では機能しないというパラダイムを大きく変化させる可能性があります。また、がん細胞やiPS細胞等ではヘテロクロマチン構造が異なっている事が示されていますので、がんの発生の理解やiPS細胞産生の理解につながります。
研究成果のポイント
● ヘテロクロマチン形成に関与するタンパク質の化学修飾「リン酸化」の役割の解明
● 乳がんの生成と転移に関与するタンパク質HP1αの構造的理解
● 教科書でよく知られているタンパク質の球状構造ではなく、細長く伸びた紐構造の役割を解明
※ 本研究の成果は、文部科学省「先端研究基盤共用・プラットフォーム形成事業」と文部科学省及び国立研究開発法人日本医療研究開発機構 創薬等ライフサイエンス研究支援基盤事業(創薬等支援技術基盤プラットフォーム事業)の支援により得られました。
● 乳がんの生成と転移に関与するタンパク質HP1αの構造的理解
● 教科書でよく知られているタンパク質の球状構造ではなく、細長く伸びた紐構造の役割を解明
※ 本研究の成果は、文部科学省「先端研究基盤共用・プラットフォーム形成事業」と文部科学省及び国立研究開発法人日本医療研究開発機構 創薬等ライフサイエンス研究支援基盤事業(創薬等支援技術基盤プラットフォーム事業)の支援により得られました。
研究の概要と成果
私達のDNAは、肝臓や心臓、神経など各々の細胞で特定の遺伝子を発現し特定の細胞機能を果たしています。ヒトでは約250種類といわれる細胞は基本的には同じDNAを持っていますが、細胞ごとにDNAの折れ畳まり方が違い、発現する遺伝子が異なっています。細胞の核内でDNAは4種類のヒストンタンパク質に巻きついた玉のような構造(ヌクレオソームという。)を取り、それが数珠状に連なりクロマチン構造を形成しています。クロマチン構造としてユークロマチンとヘテロクロマチンの2種類が知られています。ユークロマチンでは数珠状構造が緩んでいてDNAはむき出しになりその部位の遺伝子が活発に発現しているのに対し、ヘテロクロマチンではヘテロクロマチンタンパク質(HP1)によって数珠状構造が凝縮しDNAが外からは見えなくなり遺伝子の発現が抑えられます。ユークロマチンとヘテロクロマチンはヌクレオソームを構成する4種類のヒストンタンパク質のひとつであるヒストンH3の化学修飾によって区別されます。例えばヒストンH3のN末のふらふらした紐(フレキシブルテイル)の9番目のリシンというアミノ酸がメチル化されていると、HP1タンパク質が特異的に結合し凝集が起きます。HP1タンパク質に存在する球状の領域(クロモドメインという。)に特徴的なポケットがあり、そのポケットの中にメチル化されたヒストンH3の9番目のリシンが特異的に納まって、ふらふらしたヒストンの紐の一部を固定していました。しかし、2011年に、クロモドメインからふらふらと突き出た4個のアミノ酸のセリンがリン酸化されると、HP1タンパク質とメチル化されたヒストンH3の結合が強くなる事を、当時理化学研究所の発生再生研に所属していた中山博士(現名古屋市立大学)が発見しました。なぜHP1タンパク質のふらふらした紐のような領域のリン酸化がメチル化されたヒストンH3のふらふらした領域の結合を強くするのかが不明でしたが、今回、横浜市大鶴見キャンパスに設置した世界最高級感度を誇るNMR分光器を西村学長補佐のグループの元特任助教の下條氏と修士を卒業した川口氏が使用して紐の部分と球状構造を解析し、スーパーコンピュータを木寺教授のグループの森次特任准教授、博士研究員の大森氏、修士を卒業した樋口氏が使用して紐と紐の相互作用を分子動力学計算で解析し、さらに実験室内小型X線小角散乱装置を佐藤教授のグループの小田特任助教が使用して紐の長さを解析した共同研究により、HP1タンパク質のリン酸化された紐がメチル化されたヒストンH3の紐とお互いがふらふらした伸びた紐同士で結合をしている様子を明らかにしました。これは、今までタンパク質の働きは球状の構造体が重要であるという生物学の教科書で確立していたパラダイムを大きく変革する成果です。
研究内容の詳細
DNAは細胞の核の中で折り畳まれたクロマチン構造を取っています。クロマチン構造には2種類が存在し、DNAが剥き出しにあって遺伝子が活発に発現しているユークロマチンと、DNAがきつく折り畳まれたヘテロクロマチンです。ヒトには心臓や肝臓や神経など様々な機能を持った細胞があり、細胞毎に特定の遺伝子が発現しクロマチン構造が異なっています。クロマチンは4種類のヒストンタンパク質(H2A、H2B、H3、H4)が2個ずつ集まった球状のヒストン8量体(オクタマー)の周囲に146塩基対からなるDNAが巻きついたヌクレオソームが基本構造になり、数珠状につながっています。ユークロマチンではヌクレオソームの数珠状の構造が緩んでいて、数珠と数珠を剥き出しのDNAが結んでいる構造ですが、ヘテロクロマチンではヌクレオソーム同士が固まった集合体を形成しています。ユークロマチンとヘテロクロマチンの違いはヒストンの化学修飾の違いで説明できます。例えばユークロマチンのヒストンH3のN末の4番目のアミノ酸のリシンがメチル化されているのに対し、ヘテロクロマチンではヒストンH3のN末の9番目のアミノ酸のリシンがメチル化(H3K9me)されています。9番目のリシンがメチル化されたヒストンH3はヘテロクロマチンタンパク質(HP1)によって認識されHP1タンパク質がヌクレオソームを会合させます。
HP1タンパク質がヒストンH3K9meに結合する領域をクロモドメインと呼びます。HP1のクロモドメインとヒストンH3K9meの結合様式は2002年に米国のグループが明らかにしました(参考文献1)。クロモドメインは球状の構造を取り、ヒストンH3の5番目から8番目のアミノ酸がクロモドメインの溝にはまり込み9番目のメチル化リシンがポケットに納まっていました。HP1の20番から80番のアミノ酸がクロモドメインの球状構造を形成しています。ところが現在名古屋市大の中山教授のグループが2004年にクロモドメインの球状構造のN末の11番目から14番目にあるセリン残基がリン酸化されるとヒストンH3K9meとの結合が非常に強くなる事を見出しました。N末領域は球状クロモドメインから紐のように突き出ていて、その紐のリン酸化によりヒストンH3K9meの紐との結合が考えられます。タンパク質構造の教科書によると、アミノ酸の紐からなるタンパク質は機能するときは折れたたまれた球状構造で、通常天然型と呼び、温度を上げたりするとタンパク質は変性し機能がなくなることが知られています。しかし最近単独で存在するときには変性した紐状構造である天然変性タンパク質の例が知られてきました。これら天然変性タンパク質も機能するために相手の球状タンパク質と結合すると相手に合わせて紐からヘリックス等に変化する事(結合にカップルした構造形成)が知られています。私達は最近、天然変性タンパク質であるがん抑制タンパク質p53のリン酸化された転写活性化ドメインやヌクレオチド除去修復因子XPCの酸性ドメインが基本転写因子のTFIIHのp62タンパク質と結合すると紐状構造でp62の表面に固定化される例を最近報告しました(参考文献3と4)。
HP1タンパク質がヒストンH3K9meに結合する領域をクロモドメインと呼びます。HP1のクロモドメインとヒストンH3K9meの結合様式は2002年に米国のグループが明らかにしました(参考文献1)。クロモドメインは球状の構造を取り、ヒストンH3の5番目から8番目のアミノ酸がクロモドメインの溝にはまり込み9番目のメチル化リシンがポケットに納まっていました。HP1の20番から80番のアミノ酸がクロモドメインの球状構造を形成しています。ところが現在名古屋市大の中山教授のグループが2004年にクロモドメインの球状構造のN末の11番目から14番目にあるセリン残基がリン酸化されるとヒストンH3K9meとの結合が非常に強くなる事を見出しました。N末領域は球状クロモドメインから紐のように突き出ていて、その紐のリン酸化によりヒストンH3K9meの紐との結合が考えられます。タンパク質構造の教科書によると、アミノ酸の紐からなるタンパク質は機能するときは折れたたまれた球状構造で、通常天然型と呼び、温度を上げたりするとタンパク質は変性し機能がなくなることが知られています。しかし最近単独で存在するときには変性した紐状構造である天然変性タンパク質の例が知られてきました。これら天然変性タンパク質も機能するために相手の球状タンパク質と結合すると相手に合わせて紐からヘリックス等に変化する事(結合にカップルした構造形成)が知られています。私達は最近、天然変性タンパク質であるがん抑制タンパク質p53のリン酸化された転写活性化ドメインやヌクレオチド除去修復因子XPCの酸性ドメインが基本転写因子のTFIIHのp62タンパク質と結合すると紐状構造でp62の表面に固定化される例を最近報告しました(参考文献3と4)。
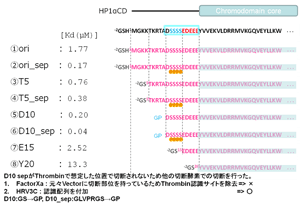
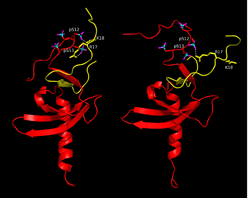
HP1クロモドメイン単独(Y20)では解離定数が13.3μMですが、N末を伸ばしてリン酸化するとD10_sep解離定数が0.04μMと330倍ヒストンH3K9meとの結合が強くなりました。N末まで伸ばすと結合が少し弱くなりますが、リン酸化すると非リン酸化に比べて約10倍結合が強くなりました。この現象は今までのクロモドメイン単独の球状構造では説明が出来なく、クロモドメインから伸びた紐がヒストンの紐と相互作用していることを意味しています(右図)。
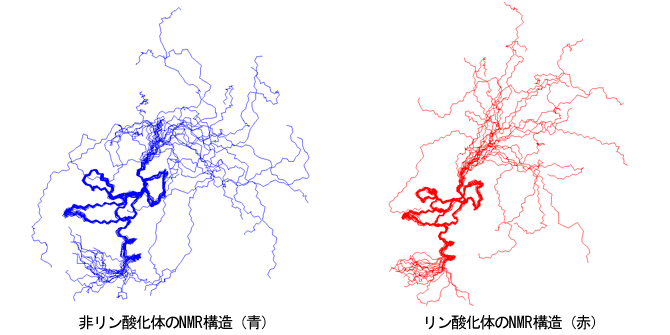
NMRでリン酸化された構造(赤)とリン酸化されていない構造(青)を決定しました、クロモドメインの領域の構造には全く差がないが、紐のダイナミクスに差がありました。リン酸化された方の紐がリン酸化されていない紐に比べてより伸びた構造でした(図1)。
図1
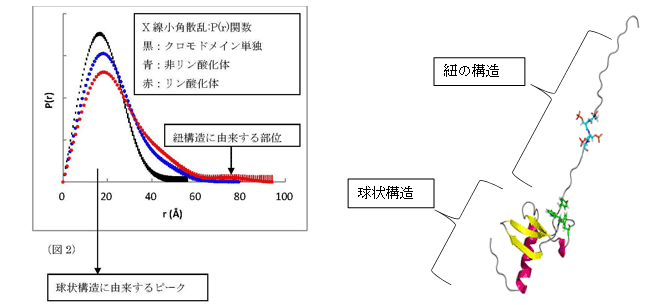
実際に紐が伸びた構造かどうかをX線小角散乱で確認しました。X線小角散乱の実験から、クロモドメイン単独では球状構造(黒)ですが、リン酸化されていない紐がついていると(青)伸びた紐構造に由来するシグナルが見えました。リン酸化体(赤)では更に紐が伸びた構造でした。溶液中のX線小角散乱の実験からも紐の挙動が解明されました(図2)。
図2
伸びた紐が実際にヒストンH3K9meの紐とどのように相互作用しているかをNMRで解析した結果、図3の(a)に示すようにHP1のリン酸化された紐の領域とヒストンH3K9meの紐の領域がふらふらとお互いに揺らいでいましたが、その中には図4に示すようにお互いが結合している構造もありました。紐と紐の結合は非常に動的で静的な安定構造ではなかったので、レプリカ交換分子動力学計算(REMD)でその挙動を調べました。REMDでも図4の右に示すように赤で示したHP1のリン酸化された紐と黄色で示したヒストンH3の紐が静電的に結合していました。このように紐と紐の相互作用をNMRと分子動力計算より、初めてその実態を解明する事が可能となりました。
図3
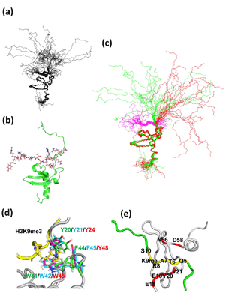
NMRで解析したリン酸化HP1とヒストンH3K9meの複合体構造の重ね書き(a)とその一つ(b)。フリー(赤)と複合体構造の比較(c)。メチル基に結合するポケット(d)。H3K9meの5番目から8番目の結合様式。
図3補足
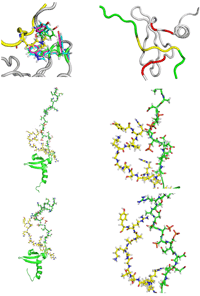
NMRで得られた複合体構造中で得られた紐と紐の結合の一例。
図4
REMD計算の結果得られた複合体構造。
HP1が赤でH3K9meが黄色で表示されている。
紐と紐の結合の一例。
HP1が赤でH3K9meが黄色で表示されている。
紐と紐の結合の一例。
今後の展開
ヘテロクロマチンタンパク質HP1は乳がんに関連する事が知られています。紐と紐の結合の実態に基づいて乳がん治療候補化合物のデザインが将来可能になります。なお、iPS細胞ではヘテロクロマチン構造を変化させる事が重要なので、ヘテロクロマチン構造形成を制御する事が可能となればiPS細胞の制御も可能になることが期待されます。さらに、今までは教科書的にはタンパク質の働きは球状構造の変化が重要だとされてきて、その静的な構造変化を結晶化して構造解析を行う事が現在のパラダイムであるが、今回の研究等により、タンパク質の紐と紐の動的な結合が働きに重要である事が分かってきたので、今後教科書的な考え方のパラダイムシフトを起こす必要があります。
参考文献
(1)Jacobs, S.A. & Khorasanizadeh, S. Structure of HP1 chromodomain bound to a lysine 9-methylated histone H3 tail. Science 295, 2080-2083 (2002).
(2)Hiragami-Hamada, K. et al. N-terminal phosphorylation of HP1α promotes its chromatin binding. Mol. Cell. Biol. 31, 1186-1200 (2011).
(3)Okuda, M. & Nishimura, Y. Extended string binding mode of the phosphorylated transactivation domain of tumor suppressor p53. J. Am. Chem. Soc. 136, 14143- 14152 (2014).
(4)Okuda, M., Kinoshita, M., Kakumu, E., Sugasawa, K. & Nishimura, Y. Structural Insight into the Mechanism of TFIIH Recognition by the Acidic String of the Nucleotide Excision Repair Factor XPC. Structure 23, 1827-1837 (2015).
(2)Hiragami-Hamada, K. et al. N-terminal phosphorylation of HP1α promotes its chromatin binding. Mol. Cell. Biol. 31, 1186-1200 (2011).
(3)Okuda, M. & Nishimura, Y. Extended string binding mode of the phosphorylated transactivation domain of tumor suppressor p53. J. Am. Chem. Soc. 136, 14143- 14152 (2014).
(4)Okuda, M., Kinoshita, M., Kakumu, E., Sugasawa, K. & Nishimura, Y. Structural Insight into the Mechanism of TFIIH Recognition by the Acidic String of the Nucleotide Excision Repair Factor XPC. Structure 23, 1827-1837 (2015).
用語説明
NMR(核磁気共鳴)
強い磁場中にタンパク質を置くとタンパク質を構成する原子核の核スピンが変化し、それを解析することによってタンパク質の構造を解析する手法である。同様の原理でMRIは強い磁場中におかれた生体中の水分子の水素原子核の核スピンをモニターしている。タンパク質の構造の詳細を知るためには水素原子核だけの情報では不十分で、さらに質量13の炭素原子(通常は質量12)や質量15の窒素原子(通常は質量14)等の安定同位体を使用する必要があり、その目的にために特別なタンパク質を調製する。横浜市立大学には世界的に最高レベルのNMR装置が文科省のプラットフォーム形成事業により整備されている。
X線小角散乱(SAXS)
溶液中に存在するタンパク質にX線を照射しその散乱を解析し溶液中に存在するタンパク質の構造を解析する。通常は播磨や筑波にある放射光施設を利用し、試料の損傷に注意しながら強いX線を照射するが、横浜市大には研究室レベルの弱いX線を照射して解析できる装置が設置されている。
レプリカ交換分子動力学計算(REMD)
計算機中でタンパク質のダイナミクスをシミュレーションする方法が、分子動力学計算(MD)である。しかしMDは、高々μ秒(10-6秒)程度の時間しか追えない。紐の動きはμ秒よりもはるかに遅い運動であり、そのような遅い時間スケールの運動を捉えるために、異なる温度でのシミュレーションを多数同時に行う(ここでは48のシミュレーションを同時実行)REMDという大きな計算資源を使う方法を用いた。
強い磁場中にタンパク質を置くとタンパク質を構成する原子核の核スピンが変化し、それを解析することによってタンパク質の構造を解析する手法である。同様の原理でMRIは強い磁場中におかれた生体中の水分子の水素原子核の核スピンをモニターしている。タンパク質の構造の詳細を知るためには水素原子核だけの情報では不十分で、さらに質量13の炭素原子(通常は質量12)や質量15の窒素原子(通常は質量14)等の安定同位体を使用する必要があり、その目的にために特別なタンパク質を調製する。横浜市立大学には世界的に最高レベルのNMR装置が文科省のプラットフォーム形成事業により整備されている。
X線小角散乱(SAXS)
溶液中に存在するタンパク質にX線を照射しその散乱を解析し溶液中に存在するタンパク質の構造を解析する。通常は播磨や筑波にある放射光施設を利用し、試料の損傷に注意しながら強いX線を照射するが、横浜市大には研究室レベルの弱いX線を照射して解析できる装置が設置されている。
レプリカ交換分子動力学計算(REMD)
計算機中でタンパク質のダイナミクスをシミュレーションする方法が、分子動力学計算(MD)である。しかしMDは、高々μ秒(10-6秒)程度の時間しか追えない。紐の動きはμ秒よりもはるかに遅い運動であり、そのような遅い時間スケールの運動を捉えるために、異なる温度でのシミュレーションを多数同時に行う(ここでは48のシミュレーションを同時実行)REMDという大きな計算資源を使う方法を用いた。
論文著者ならびにタイトルなど
<論文著者>
Hideaki Shimojo1, Ayumi Kawaguchi1, Takashi Oda1, Nobuto Hashiguchi1, Satoshi Omori1, Kei Moritsugu1, Akinori Kidera1, Kyoko Hiragami-Hamada2, Jun-ichi Nakayama3, Mamoru Sato1 and Yoshifumi Nishimura1*
1 Graduate School of Medical Life Science, Yokohama City University, 1-7-29 Suehiro-cho, Tsurumi-ku, Yokohama, Kanagawa 230-0045, Japan
2 Division of Genome Technologies, RIKEN Center for Life Science Technologies, 1-7-22 Suehiro-cho, Tsurumi-ku, Yokohama, Kanagawa 230-0045, Japan
3 Graduate School of Natural Sciences, Nagoya City University, 1 Yamanohata, Mizuho, Nagoya, Aichi 467-8501, Japan
<タイトル>
Extended string-like binding of the phosphorylated HP1α N-terminal tail to the lysine 9-methylated histone H3 tail
Hideaki Shimojo1, Ayumi Kawaguchi1, Takashi Oda1, Nobuto Hashiguchi1, Satoshi Omori1, Kei Moritsugu1, Akinori Kidera1, Kyoko Hiragami-Hamada2, Jun-ichi Nakayama3, Mamoru Sato1 and Yoshifumi Nishimura1*
1 Graduate School of Medical Life Science, Yokohama City University, 1-7-29 Suehiro-cho, Tsurumi-ku, Yokohama, Kanagawa 230-0045, Japan
2 Division of Genome Technologies, RIKEN Center for Life Science Technologies, 1-7-22 Suehiro-cho, Tsurumi-ku, Yokohama, Kanagawa 230-0045, Japan
3 Graduate School of Natural Sciences, Nagoya City University, 1 Yamanohata, Mizuho, Nagoya, Aichi 467-8501, Japan
<タイトル>
Extended string-like binding of the phosphorylated HP1α N-terminal tail to the lysine 9-methylated histone H3 tail
お問い合わせ先
本資料の内容に関するお問合せ
公立大学法人横浜市立大学大学院生命医科学研究科
学長補佐 西村 善文
横浜市鶴見区末広町1-7-29
Tel:045-508-7211/7212
E-mail: (送信の際に、※を@にご変更ください。)
学長補佐 西村 善文
横浜市鶴見区末広町1-7-29
Tel:045-508-7211/7212
E-mail: (送信の際に、※を@にご変更ください。)
取材対応窓口、資料請求など
公立大学法人横浜市立大学 研究推進課長 竹内 紀充
Tel:045-787-2019
E-mail:
Tel:045-787-2019
E-mail: