2021.11.30
- TOPICS
- 大学
- 学生の活躍
本学卒業生で生命医科学研究科 特任助手の柴橋 佑希さんが、情報計算科学生物(CBI)学会2021年大会でエクセレントポスター賞を受賞しました。
本学生命医科学研究科 博士前期課程の卒業生で、生命医科学研究科 特任助手 の柴橋 佑希さん(所属研究室:池口 満徳教授)は、2021年10月26日(火)〜28日(木)に開催された情報計算科学生物(CBI)学会2021年大会においてポスター発表を行い、エクセレントポスター賞を受賞しました。
受賞者
生命医科学研究科 特任助手
柴橋 佑希(しばはし ゆうき)さん
発表演題
「Inhibitor discovery targeting UHRF1 by MD simulations and biochemical analyses」
(分子動力学シミュレーションと生化学的解析によるUHRF1を標的とした阻害剤探索)
受賞者
生命医科学研究科 特任助手
柴橋 佑希(しばはし ゆうき)さん
発表演題
「Inhibitor discovery targeting UHRF1 by MD simulations and biochemical analyses」
(分子動力学シミュレーションと生化学的解析によるUHRF1を標的とした阻害剤探索)

柴橋 佑希さんのコメント
この度は栄誉ある賞をいただき、大変光栄に存じます。発表を行うにあたり、ご協力を賜りました池口教授、及び生命情報科学研究室の皆様、そして共同研究者としてお力添えをいただきました構造生物学研究室の皆様に深く感謝を申し上げます。当学会では、多くの先生と議論を行い、非常に有意義な時間を過ごすことができました。今回の発表を通じて得た学びを活かして、より研究に励んで参りたいと思います。
指導教員 池口 満徳 教授のコメント
柴橋さんが、大学院生時代から行ってきた研究が実を結びました。投稿論文も採択され、大きな成果だと思います。実験研究者との共同研究もうまく進み、たいへんよかったように思います。今後においても、今回の成果はとても活きるように思いますので、ぜひ精進して欲しいと思います。
発表内容
我々の身体を構成する様々な細胞は、種類毎に固有の形質を持っています。この形質を決定付けるのがDNAメチル化です。細胞が分化してDNAが複製される際に、DNAメチル化情報は、DNA維持メチル化機構によって受け継がれます。DNAメチル化や、DNA維持メチル化の異常は、がんの要因であることが知られています。例えば、がん抑制遺伝子が過剰にメチル化された状態が、DNA維持メチル化機構によって継承されてしまうと、がん細胞が増殖し続けてしまいます。本研究で着目したUHRF1タンパク質はDNA維持メチル化を制御する因子で、様々ながん細胞で過剰に発現していることが報告されています。そのため、UHRF1の働きを阻害する薬ができれば、異常なDNAメチル化の維持を阻害できるので、がん治療につながると期待されています。UHRF1には、tandem tudor domain(TTD)と呼ばれるドメインがあり、TTD中のペプチド結合溝にLIG1分子が結合し機能を調整することが明らかになっています。そこで、本研究ではTTDの結合溝にはまり、機能を阻害する医薬品候補化合物を探索しました。20万個程度の候補化合物を全て実験することは不可能ですが、インシリコスクリーニング技術を駆使することで、実験可能な100個程度まで絞り込むことができました。従来の、静的な構造を使ったドッキングシミュレーションだけでなく、分子の動きまで考慮できる分子動力学シミュレーションをスクリーニングサイクルに組み込み、候補化合物がどれだけ安定に結合できるかも検証しました。その結果、UHRF1に結合するヒット化合物を得ることに成功しました。実験結果を振り返ってみると、分子動力学シミュレーションの指標が効果的にランキングできていることがわかりました。今後は、得られたヒット化合物を育て、より結合能が強い化合物を探索したいと思います。
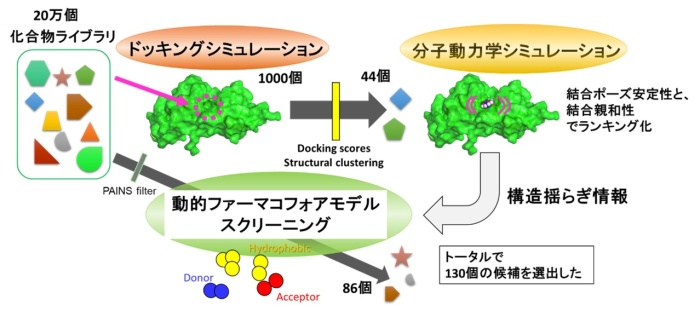
The accumulation of epigenetic alternation is one of the major causes of tumorigenesis. Because aberrant DNA methylation patterns cause genome instability and silencing of tumor suppressor genes in various types of tumor, drug discovery targeting the DNA methylation-regulating factors has a potential to develop a cancer treatment drug. In mammals, ubiquitin-like containing PHD and RING finger domain 1 (UHRF1) plays an essential factor for DNA methylation maintenance [1,2]. In addition, UHRF1 is overexpressed in various cancer cells, and down-regulation of UHRF1 reactivates the expression of tumor suppressor genes, suggesting inhibitor of UHRF1 is promising for drugs. UHRF1 consists of multi domains. In particular, the peptide binding groove in tandem Tudor domain (TTD-groove) functions as a binding platform for inter- or intra-molecular interactions, and the recruitment of UHRF1 to replication sites is triggered by the interaction of DNA ligase 1 di/trimethylated on Lys126 (LIG1K126me3) to the TTD-groove. Therefore, we focused on inhibiting the interaction between TTD-groove and LIG1K126me3. To find candidate inhibitors, in-silico high-throughput screening cycle was developed and performed using over 200,000 compounds. This cycle consisted of docking simulations, molecular dynamics (MD) simulations, binding free-energy calculations, and dynamical pharmacophore screening. The binding affinity was evaluated by the docking score, MM-GBSA score, and the binding free energy, and the pose stability was evaluated by RMSD using three independent all-atom 10-ns MD simulations. Because it is generally not known which of the computational scores and/or indicators in the in-silico scheme correlate with the affinity evaluated experimentally, the top-ranked compounds were selected from multiple scores and indicators. As a results, we selected 130 candidates by this cycle, and succeeded in obtaining two hit compounds confirmed by experimental thermal stability assay and isothermal titration calorimetry. The hit compound was the top performer in three of the four indicators, i.e., all except for the docking score. In particular, the binding pose of the hit compound was quite stable in the 10-ns MD simulation. As a validation, we solved a crystal structure of the hit compound bound UHRF1 complex. The binding pose in the crystal structure was in good agreement with that observed in the MD simulations. In retrospective evaluation, the two hit compounds were evaluated as promising by MD-based metrics, and the hit rate among the compounds was 10.5%, which was higher than a commonly mentioned rate, 1%, indicating that MD simulation was useful to screen the compounds which bind to the target protein.
[1] Sharif, J.; et al., The SRA Protein Np95 Mediates Epigenetic Inheritance by Recruiting Dnmt1 to Methylated DNA, Nature, 2007, 450, 908-912.
[2] Bostick, M.; et al., UHRF1 Plays a Role in Maintaining DNA Methylation in Mammalian Cells, Science, 2007, 317, 1760-1764.
[1] Sharif, J.; et al., The SRA Protein Np95 Mediates Epigenetic Inheritance by Recruiting Dnmt1 to Methylated DNA, Nature, 2007, 450, 908-912.
[2] Bostick, M.; et al., UHRF1 Plays a Role in Maintaining DNA Methylation in Mammalian Cells, Science, 2007, 317, 1760-1764.