2021.11.30
- TOPICS
- 大学
- 学生の活躍
- 理学部
国際総合学部4年・中居 雪菜さんが、学部生ながら情報計算科学生物(CBI)学会2021年大会でエクセレントポスター賞を受賞しました。
国際総合学部理学系生命医科学コース 4年の中居 雪菜さん(指導教員:池口 満徳教授)は、2021年10月26日(火)〜28日(木)にオンラインで開催された情報計算科学生物(CBI)学会2021年大会においてポスター発表を行い、エクセレントポスター賞を受賞しました。
受賞者
国際総合科学部 4年
中居 雪菜(なかい ゆきな)さん
発表演題
「Activation Pathway of IL-2-inducible T-cell kinase Explored by Tree-Search Molecular Dynamics」
(ツリーサーチ分子動力学法によるIL-2誘導型T細胞キナーゼの活性化経路の解明)
受賞者
国際総合科学部 4年
中居 雪菜(なかい ゆきな)さん
発表演題
「Activation Pathway of IL-2-inducible T-cell kinase Explored by Tree-Search Molecular Dynamics」
(ツリーサーチ分子動力学法によるIL-2誘導型T細胞キナーゼの活性化経路の解明)

中居 雪菜さんのコメント
この度は名誉ある賞をいただき、大変光栄に思っております。研究や発表準備にあたり、親切にご指導いただきました、池口教授をはじめ共同研究者の皆様に、心より感謝申し上げます。発表当日は、様々な専門家の方々にご質問やアドバイスをいただき、自身の未熟さを改めて痛感しました。本学会での貴重な経験を活かし、より研究活動に精進していきたいと思います。
指導教員 池口 満徳 教授のコメント
学部4年生の段階で、学会で発表し、賞までいただけたのは、すばらしい成果だと思います。いろいろと工夫しながら、研究を行ってきた成果だと思います。今後、大学院に進み、いろいろと問題にもぶち当たるとは思いますが、ぜひそれを乗り越えて、一段と研究を発展させてほしいと思います。
発表内容
Interleukin-2-inducible T-cell Kinase (ITK)は、T細胞受容体のシグナル伝達を担う非受容体型チロシンキナーゼであり、その阻害薬はアレルギーの治療薬になり得ると期待されています。プロテインキナーゼは、現在上市されている医薬品の多くが標的としている代表的なタンパク質で、不活性型と活性型の2つの構造をとることが知られています。ITKについても、その2つの状態の結晶構造は解かれていますが、不活性型/活性型の2状態間をつなぐ構造変化パスウェイや、その活性化メカニズムについては明らかになっておらず、それらについての詳細な理解が求められていました。特に、別のキナーゼタンパク質では、中間状態構造の存在も示唆されており、ITKではどうなのか興味がもたれています。一般的に、タンパク質の構造変化の情報は、分子動力学シミュレーションによって得ることができます。しかし、状態間の構造変化はダイナミックであり、通常の分子動力学シミュレーションで追跡できるタイムスケールを超えてしまいます。そこで、本研究では、AlphaGoでも用いられている木探索アルゴリズムをベースにして、分子動力学シミュレーションと組み合わせた、ツリーサーチ分子動力学法によって構造変化のパスウェイ探索を行いました。特徴的なヘリックスとループの構造に対して、評価指標を作成し、構造変化が起こる方向へ構造探索が進むようにしました。ツリーサーチでは、構造探索が無理な方向に進むと、過去の構造に戻って再探索する仕組みを導入することで、効率的に探索できるようにしました。その結果、活性型構造から、中間的な構造を経由して、不活性型の構造に至る構造変化パスウェイを得ることに成功しました。今後は、得られたパスウェイ上の構造からスーパーコンピュータ富岳等を使って分子動力学シミュレーションを実行し、構造変化途中の構造安定性まで評価してみたいと思います。
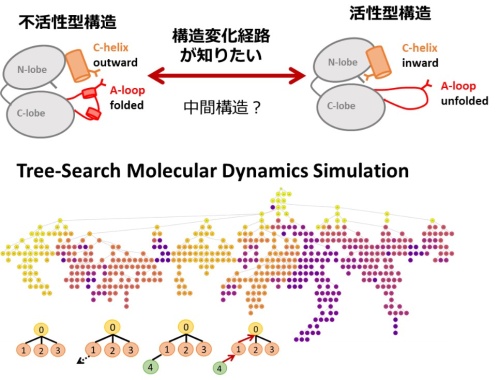
Interleukin-2 (IL-2) -inducible T cell kinase (ITK) is a non-receptor tyrosine kinase that plays an essential role in T cell receptor signaling [1]. Because ITK also mediates the secretion of Th2 cytokines, including IL-4, IL-5, and IL-13, and regulates the development of effective Th2 responses during allergic asthma and parasitic infections, ITK is considered as a potential therapeutic target for the treatment of the Th2-mediated inflammatory diseases [1]. Several crystal structures of the kinase domain of human ITK in the inactive and active states have been solved so far [1], and show the structural features in the two endpoint states: the unfolded/folded conformations of A-loop and the inward/outward positions of αC-helix. However, the crystal structure alone does not provide the details of the activation mechanism coupled with the conformational changes between the endpoint states. In particular, the structural information of the intermediate state is necessary to understand the activation mechanism structurally. To understand the activation mechanism of ITK, we studied conformational changes between the inactive and active states using all-atom molecular dynamics (MD) simulations. Because the time scale of the conformational change underlying the activation event exceeds the conventional MD simulations, an enhanced pathway-search method based on MD and a tree-search algorithm, termed the Tree-Search Molecular Dynamics simulation (TS-MD) [2] was employed for the pathway search during the activation event. We constructed evaluation functions that can describe the structural changes of αC-helix and A-loop, respectively, and conducted multiple rounds of TS-MD. First, we conducted TS-MD from the active conformation using an evaluation function that represents only the conformational change of αC-helix, and succeeded in obtaining the intermediate structure and the pathway to the intermediate structure, which has the structural features reported for other kinases. Second, TS-MD from the intermediate structure was conducted using an evaluation function that represents only the conformational change of A-loop. As a result, we succeeded in obtaining a pathway leading to a structure close to the inactive conformation. In conclusion, using TS-MD, the conformational changes during the activation event of ITK were successfully sampled.
[1] Kutach AK et al. Crystal structures of IL-2-inducible T cell kinase complexed with inhibitors: insights into rational drug design and activity regulation: crystal structures of ITK-inhibitor complexes. Chemical Biology & Drug Design. 2010, 76(2), 154-163.
[2] Shin K et al. Enhancing Biomolecular Sampling with Reinforcement Learning: A Tree Search Molecular Dynamics Simulation Method. ACS Omega. 2019, 4(9), 13853-13862.
[1] Kutach AK et al. Crystal structures of IL-2-inducible T cell kinase complexed with inhibitors: insights into rational drug design and activity regulation: crystal structures of ITK-inhibitor complexes. Chemical Biology & Drug Design. 2010, 76(2), 154-163.
[2] Shin K et al. Enhancing Biomolecular Sampling with Reinforcement Learning: A Tree Search Molecular Dynamics Simulation Method. ACS Omega. 2019, 4(9), 13853-13862.