2009.04.01
- プレスリリース
- 研究
生命ナノシステム科学研究科生体超分子システム科学専攻の片平正人教授のグループ(古川亜矢子博士研究員、永田崇助教)は、抗エイズウィルス活性を有するヒトのタンパク質APOBEC3Gに関し、立体構造及び標的DNAとの相互作用様式をNMR(核磁気共鳴)法によって決定し、さらに酵素反応をNMRシグナルを用いてリアルタイムでモニタリングする事に成功しました。この研究は千葉工業大学・高久洋教授のグループ及び理化学研究所の横山茂之領域長のグループとの共同研究による成果です。
研究概要
エイズウィルスHIVの感染に対する防衛機構として、ヒトはAPOBEC3Gというタンパク質を有しています。ヒトの細胞に侵入したHIVは、自分のゲノム情報を含むRNAを鋳型として、それと相補的なDNA(マイナス鎖DNA)を一端合成し、その後これを鋳型として再度相補的なDNA(プラス鎖DNA)を合成します。こうしてウィルスのゲノム情報を2重鎖DNAとしてヒトのDNAに組み込みます。APOBEC3Gはマイナス鎖DNA上のシトシン塩基に作用してデアミネーション反応(アミノ基NH2を除去する反応)を起こし、ウラシル塩基に変換する活性を有します。変換を受けたマイナス鎖DNAから合成されたプラス鎖DNAにおいては、HIVのゲノム情報は乱された意味の無いものとなります。この様にしてAPOBEC3Gタンパク質は、HIVの感染に対する防衛機構を担っています。
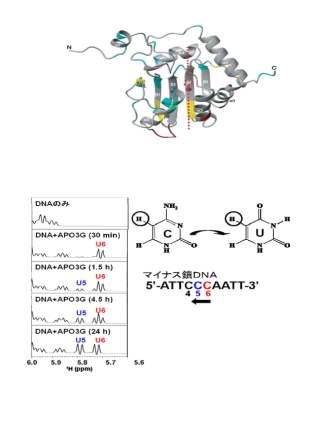
今回片平正人教授らのグループは、APOBEC3Gタンパク質の立体構造及び標的であるマイナス鎖DNAとの相互作用様式を、NMR法によって決定しました。これによりシトシン塩基がデアミネーション反応を受ける現場を視覚的に捉える事ができる様になり、APOBEC3Gの作用機構に関する理解が深まりました。更にNMRのシグナルを用いる事で、APOBEC3Gによるデアミネーション反応をリアルタイムでモニタリングする事に初めて成功しました。そしてデアミネーション反応が、マイナス鎖DNA上において3’→5’の順序をもって進行する事を見出しました。立体構造という基盤に加えて、反応のダイナミクスをリアルタイムで解明できた事により、APOBEC3Gの動作機構ひいては抗エイズウィルス活性に関して深い理解が得られました。ウィルスの有するVifというタンパク質が働くとAPOBEC3Gが抗エイズウィルス活性を十分に発揮できなくなってしまう事が、現在解決すべき課題となっています。今回得られた知見に基づいてこの課題を克服し、エイズの治療に結びつけられる事が今後期待されます。
今回片平正人教授らのグループは、APOBEC3Gタンパク質の立体構造及び標的であるマイナス鎖DNAとの相互作用様式を、NMR法によって決定しました。これによりシトシン塩基がデアミネーション反応を受ける現場を視覚的に捉える事ができる様になり、APOBEC3Gの作用機構に関する理解が深まりました。更にNMRのシグナルを用いる事で、APOBEC3Gによるデアミネーション反応をリアルタイムでモニタリングする事に初めて成功しました。そしてデアミネーション反応が、マイナス鎖DNA上において3’→5’の順序をもって進行する事を見出しました。立体構造という基盤に加えて、反応のダイナミクスをリアルタイムで解明できた事により、APOBEC3Gの動作機構ひいては抗エイズウィルス活性に関して深い理解が得られました。ウィルスの有するVifというタンパク質が働くとAPOBEC3Gが抗エイズウィルス活性を十分に発揮できなくなってしまう事が、現在解決すべき課題となっています。今回得られた知見に基づいてこの課題を克服し、エイズの治療に結びつけられる事が今後期待されます。
図 (上)NMR法によって決定されたAPOBEC3Gタンパク質の立体構造。マイナス鎖DNAとの相互作用部位に色をつけてある。予想されるマイナス鎖DNAの配置を点線で示してある。(下)APOBEC3Gタンパク質によるマイナス鎖DNA上のシトシンからウラシルへの変換のリアルタイムモニタリング。○印のNMRシグナルを時間を追って検出する事で、DNA鎖上3’→5’の順序でC→Uへの変換が起こる様子をリアルタイムで捉える事に成功した。